Ubiquitin ist ein kleines, hoch konserviertes Protein mit 76 Aminosäuren, das in Eukaryoten eine zentrale Rolle bei der zellulären Proteinregulation spielt.
"Hoch konserviert heißt, dass sich die Aminosäure-Sequenz des Proteins über die Jahrmillionen der Evolution fast nicht verändert hat. "Das Ubiquitin der Hefe und des Menschen unterscheiden sich nur in drei von 76 Resten". [3]
Es dient als molekulares Etikett, das an andere Proteine angehängt wird, um deren Schicksal innerhalb der Zelle zu bestimmen – insbesondere den Abbau durch die Proteasomen, großen Proteinkomplexen, die beschädigte, falsch gefaltete oder überflüssige Proteine erkennen und hydrolytisch in kleinere Peptide spalten.
Die Markierung erfolgt durch einen Prozess namens Ubiquitinierung: Ubiquitin mit Hilfe von drei Enzymen, der einfach als E1, E2 und E3 bezeichnet werden, kovalent an Lysinreste des Zielproteins gebunden (Einzelheiten siehe weiter unten). Mehrere Ubiquitin-Moleküle können dabei zu Polyubiquitin-Ketten verknüpft werden, was oft ein Signal für den Abbau durch die Proteasomen darstellt.
Neben dem Proteinabbau ist Ubiquitin auch an anderen zellulären Prozessen beteiligt, darunter DNA-Reparatur, Endocytose und Histonregulierung [1].
Das Ubiquitin-Molekül
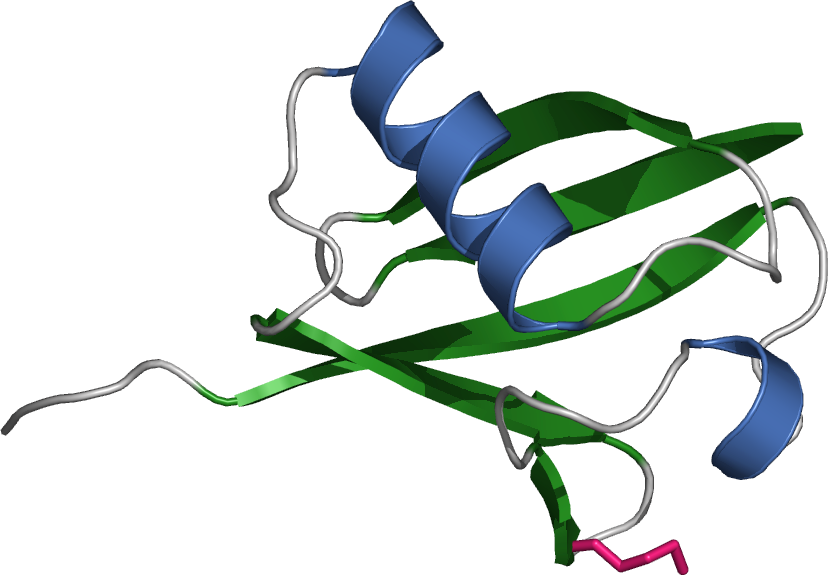
Bändermodell des Ubiquitins
en:User:Rogerdodd, Public domain, via Wikimedia Commons
Das Molekül ist mit nur 76 Aminosäuren äußerst kompakt, von einem Protein darf man eigentlich gar nicht sprechen, korrekter wäre die Bezeichnung Polypeptid (von einem Protein spricht man ja erst, wenn das Peptid mindestens 100 Aminosäuren umfasst).
Wie man auf dem Bild gut erkennen kann, besteht das Molekül aus fünf β-Faltblättern und einer α-Helix, die zentral zwischen den β-Faltblättern liegt.
Eine wichtige Bedeutung hat die Glycin-Einheit Glycin-76 am C-terminalen Ende des Peptids. Glycin-76 ist entscheidend für die kovalente Bindung des Ubiquitin-Moleküls an Lysinreste anderer Proteine, wobei das auch andere Ubiquitin-Moleküle sein können.
Diese Bindung erfolgt über eine Isopeptidbindung zwischen der Carboxy-Gruppe von Gly-76 und Aminogruppe einer Lysin-Seitenkette.
Für die Bildung von Ubiquitin-Ketten sind mehrere Lysin-Seitenketten im Ubiquitin-Molekül verantwortlich, vor allem die Lysin-Reste an den Positionen 48 und 63 sind wichtig. An diese Lysin-Seitenketten kann nämlich ein anderes Ubiquitin-Molekül mit seinem Glycin-76 kovalent ankoppeln, so dass sich Dimere, Trimere oder lange Ketten aus Ubiquitin-Molekülen bilden können.
Einfach markierte Proteine
Laut [1] spielen Proteine, die mit nur einem Ubiquitin-Molekül markiert sind, eine Rolle bei der Histonregulierung, und mit mehreren Ubiquitin-Molekülen markierte Proteine (ohne Polyubiquitin-Kette!) spielen eine Rolle bei der Endocytose.
Bildung von Ubiquitin-Ketten
Bei der Bildung von Ubiquitin-Ketten spielen drei Enzyme eine wichtige Rolle [1, 2, 3].
1. Enzym E1 (Ubiquitin aktivierendes Enzym)
Das erste Enzym E1 (Ubiquitin aktivierendes Enzym) aktiviert Ubiquitin durch Übertragung eines ATP-Moleküls. Es entsteht das Zwischenprodukt Ubiquitin-AMP:
Ub + ATP ⇌ Ub-cAMP + PPi
Dabei wird der Pyrophosphatrest freigesetzt. Im nächsten Schritt wird das Ubiquitin auf das Enzym E1 übertragen:
Ub-cAMP + E1 ⇌ E1-Ub + AMP
Am Ende von Schritt 1 ist das Ubiquitin kovalent an E1 gebunden.
2. Enzym E2 (Ubiquitin konjugierendes Enzym)
Das Enzym E2 dockt nun an den E1-Ubiquitin-Komplex an. Dann wird das Ubiquitin von E1 auf einen Cysteinrest im Enzym E2 übertragen:
E1-Ub + E2 ⇌ E2-Ub + E1
E1 steht wieder für den ersten Schritt - Aktivierung von Ub - zur Verfügung.
3. Enzym E3 (Ubiquitin-Ligase)
Das Enzym E3 (Ubiquitin-Ligase) hat nun eine zentrale Funktion. Es überträgt nämlich das Ubiquitin-Molekül von E2 auf das Zielprotein. Das wollen wir uns einmal mit einer kleinen Skizze klarmachen. Zunächst schauen wir uns das Enzym Ubiquitin-Ligase näher an:
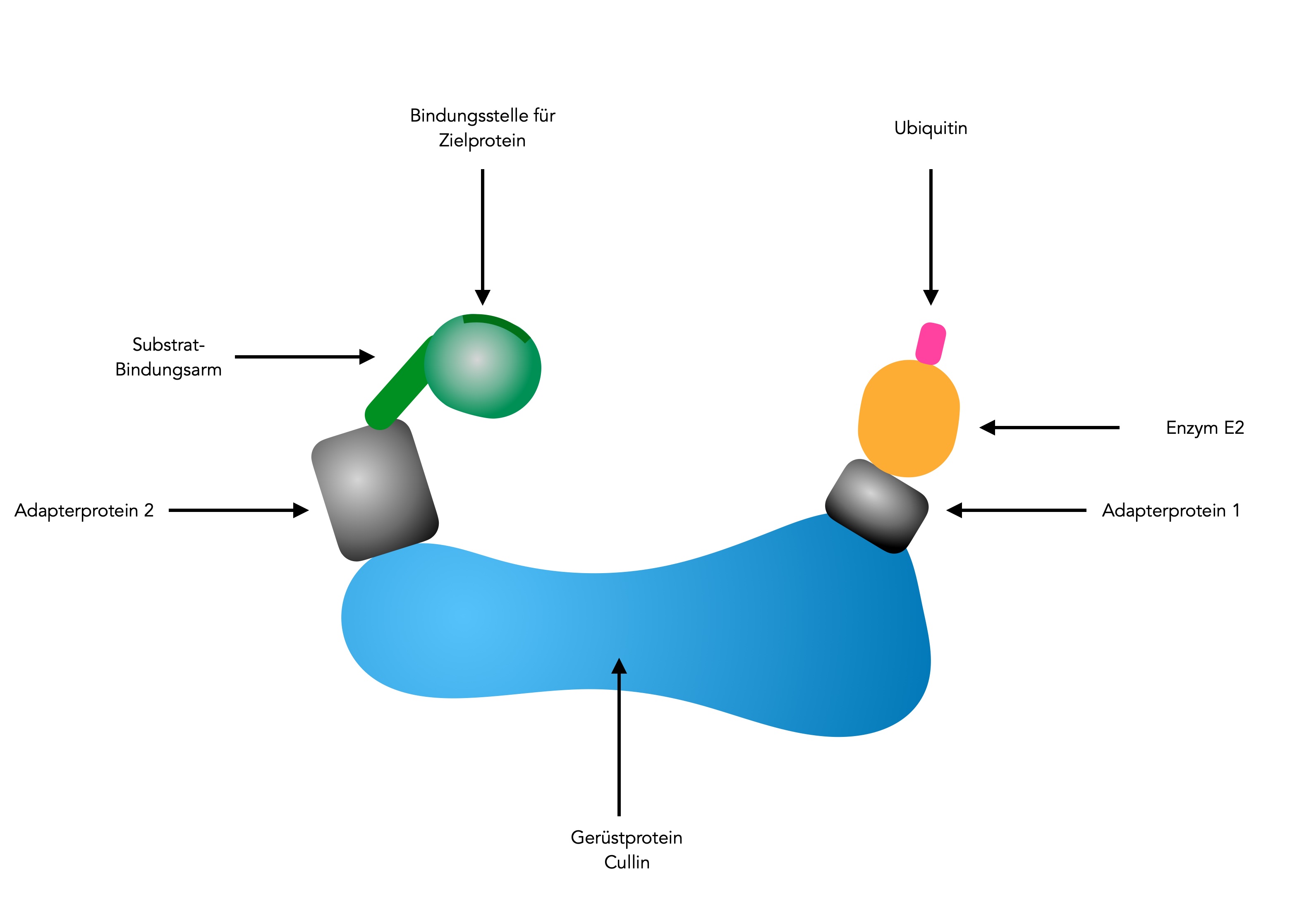
Struktur der Ubiquitin-Ligase (E3)
Autor: Ulrich Helmich 05/2025, Lizenz: Public domain
Dieses Bild zeigt den Aufbau des Enzyms aus vier Untereinheiten [1]
Die größte dieser Untereinheiten, das Cullin, kann als Gerüstprotein für E3 bezeichnet werden. Der Substratbindungsarm bindet an das Zielprotein, auf das Ubiquitin übertragen werden soll. Das Adapterprotein 2 verbindet das Gerüstprotein mit diesem Substratbindungsarm. Das Adapterprotein 1 dagegen dient zur Verbindung von E3 mit dem Enzym E2. Das Enzym E2 wiederum trägt ein Ubiquitin-Molekül.
Schauen wir uns nun an, wie das Ubiquitin von E2 auf das Zielprotein übertragen wird [1, 2, 3]:
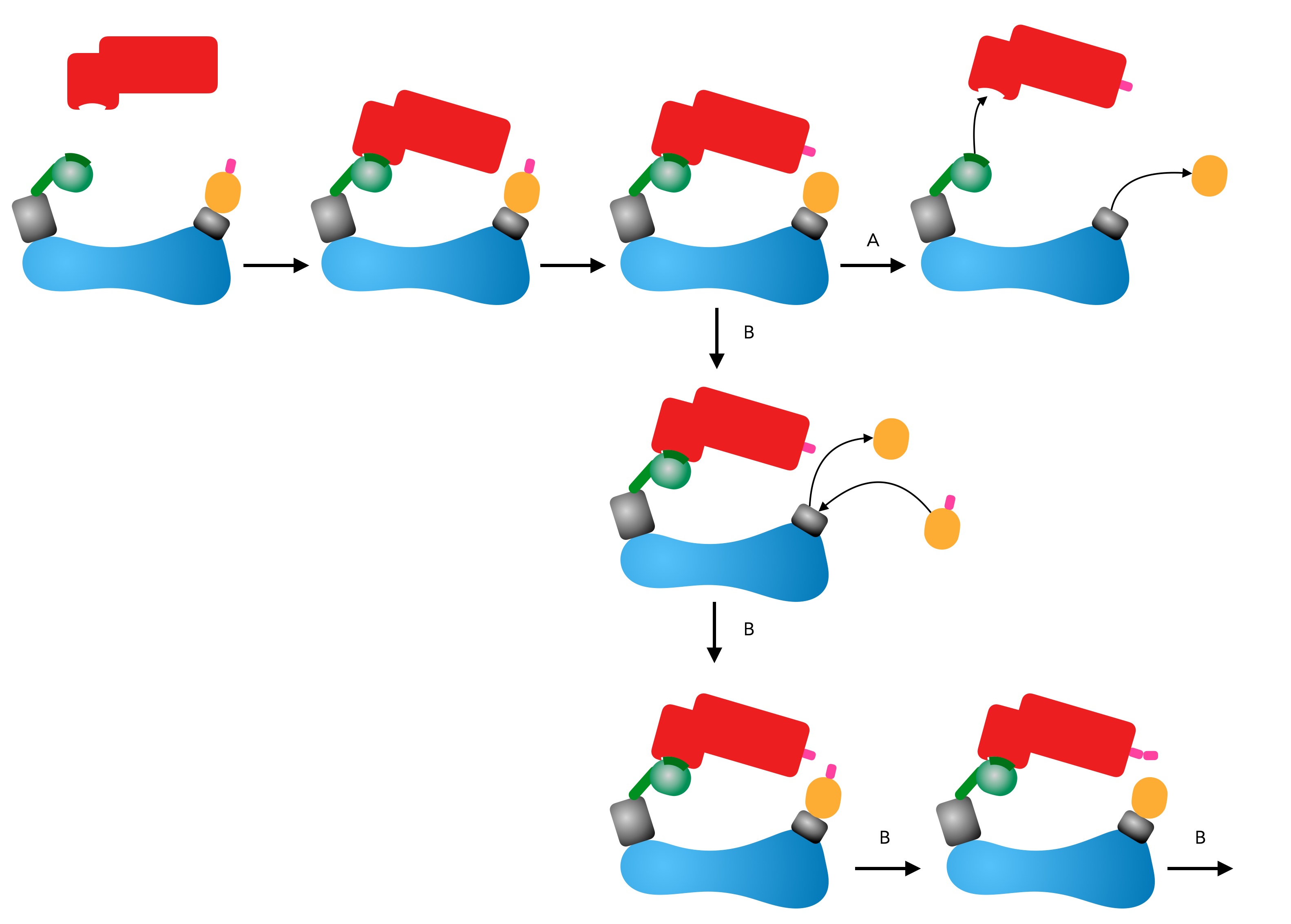
Die Arbeitsweise der Ubiquitin-Ligase
Autor: Ulrich Helmich 05/2025, Lizenz: Public domain
In der oberen Reihe sieht man den Vorgang der Ubiquitin-Übertragung in vier Einzelbildern.
Im ersten Schritt dockt das Zielprotein an den Substratbindungsarm von E3 an. Im zweiten Schritt wird das Ubiquitin-Molekül von E2 auf das Zielproteinübertragen, und in dritten Schritt, hier mit 'A' gekennzeichnet, lösen sich das Zielprotein und das E2 von der Ubiquitin-Ligase E3. Das Zielprotein ist jetzt mit einem Ubiquitin-Molekül markiert.
Ein alternativer Weg ist mit 'B' gekennzeichnet. Nach dem Transfer von Ubiquitin auf das Zielprotein bleibt dieses an E3 gebunden. Das freie E2 löst sich von dem Komplex, und ein mit Ubiquitin beladenes E2 setzt sich an das Adapterprotein 1.
Das mit E2 verbundene Ubiquitin wird dann auf das erste Ubiqutin-Molekül am Zielprotein übertragen, so dass dieses jetzt mit zwei Ubiquitin-Molekülen markiert ist. Der Pfeil am Ende soll andeuten, dass dieser Vorgang mehrmals wiederholt werden kann, so dass das Zielprotein mit drei, vier oder mehr Ubiquitin-Molekülen markiert werden kann
Bei dieser Bildung von Ubiquitin-Ketten gibt es mehrere Möglichkeiten, von denen zwei besonders wichtig sind.
- Wenn das jeweils nächste Ubiquitin-Molekül an einen Lysin-48-Rest im Vorgänger-Molekül gebunden wird, entsteht eine Ubiquitin-Kette, die als Signal für den Proteinabbau über die Proteasomen dient.
- Wird das nächste Ubiquitin-Molekül dagegen immer an einen Lysin-63-Rest im Vorgänger-Molekül gebunden, bildet sich eine Ubiquitin-Kette, die das Zielprotein so markiert, dass es für Vorgänge der DNA-Reparatur eingesetzt werden kann [1].
Quellen:
- Bruce Alberts et al. Molekularbiologie der Zelle, 7. Auflage, Weinheim 2025.
- Nelson, Cox. LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.