Die beiden DNA-Fäden eines typischen menschlichen Chromosoms sind jeweils bis zu 4 cm lang - wenn man sie vollständig auseinanderziehen würde. Ein Metaphase-Chromosom ist aber nur 1/100 Millimeter lang; es ist also 4000 mal kürzer als die beiden darin enthaltenen DNA-Fäden! Der Grund hierfür ist recht simpel: Die langen DNA-Fäden müssen in den recht kleinen Zellkern der Eukaryotenzelle hineinpassen.
Wie kommt diese starke Kondensierungder DNA (Verkürzung, Verdichtung) zustande?
Der Vergleich mit dem Aufwickeln eines Bindfadens ist nicht sehr hilfreich. Einen 3 m langen Bindfaden kann man vielleicht zu einem Knäuel mit 2 cm Durchmesser aufwickeln. Der Verkürzungsfaktor wäre dann 300:2 oder 150:1 - kein Vergleich mit der DNA, die ja einen Verkürzungsfaktor von 4000:1 (oder mehr) hat.
Die Kondensierung der DNA geschieht - im Gegensatz zum Aufwickeln eines Bindfadens - in mehreren Schritten.
Wenn Sie Schüler(inn) eines Biologie-Grundkurses sind, sollte die Kurzfassung dieser Seite eigentlich völlig ausreichen. Die lange Fassung hier ist eher für engagierte LK-Schüler(innen) oder Studienanfänger(innen) der Fächer Biologie, Medizin oder verwandter Fachbereiche geeignet.
Stufe 1: Nucleosomen

Organisation eines Nucleosoms
Autor: Ulrich Helmich, Lizenz: Lizenz: Creative Commons Attribution-ShareAlike 4.0.
Auf diesem Bild sehen wir ein sogenanntes Nucleosom. Ein Nucleosom besteht aus neuen Proteinen, von denen sich acht zu einem Komplex zusammengeschlossen haben, um den sich eine DNA-Doppelhelix ca. 1,6 bis 1,7 mal herum windet, was 146 bis 147 Basenpaaren entspricht.
Der Proteinkomplex wird aus den Histonen H2A, H2B, H3 und H4 gebildet; jedes Protein kommt zweimal in dem Komplex vor. Histone sind Proteine mit vielen Lysin- und Arginin-Resten. Vor allem die Lysin-Reste tragen oft eine positiv geladene NH3+-Gruppe in ihrer Seitenkette. Diese positiven Seitenketten ziehen dann die negativ geladenen Phosphatreste der DNA-Doppelhelix elektrisch an.
Das Histon H1, im Bild grün dargestellt, gehört nicht zu dem Proteinkomplex, sondern liegt dem Nucleosom quasi von außen auf. Das Histon H1 spielt eine wichtige Rolle bei der Verknüpfung der einzelnen Nucleosomen zur nächsthöheren Struktur.
Durch das "Wickeln" der DNA um den Histonkomplex wird ein Kondensationsfaktor von ca. 5 bis 10 erreicht. Das ist noch nicht sehr viel, wenn man an den tatsächlichen Faktor von 4000 denkt. Aber wir sind ja auch noch lange nicht fertig mit dem Kondensieren…
Auf dieser Lexikonseite finden Sie weitere Informationen zu den Nucleosomen.
Stufe 2: 10nm-Faser
Die einzelnen Nucleosomen werden durch ein ca. 50 bis 70 Basenpaare langes Stück DNA verbunden, die als Linker-DNA bezeichnet wird.
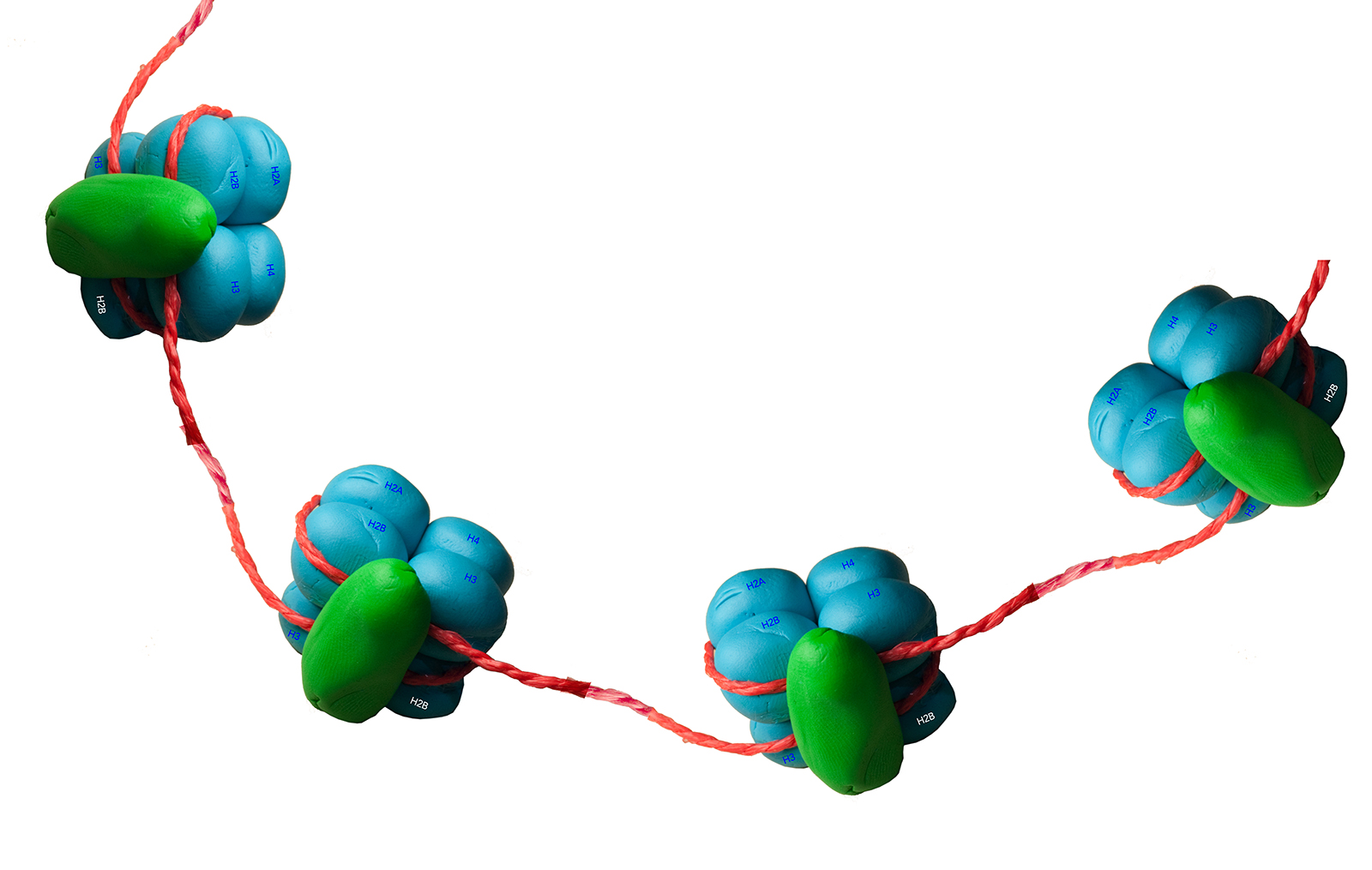
Ein Komplex aus vier Nucleosomen, die durch Linker-DNA miteinander verbunden sind.
Autor: Ulrich Helmich 05/2024, Lizens siehe Seitenende.
In Schulbüchern und auch oft in der Fachliteratur wird diese 10nm-Faser mit einer Perlenkette verglichen. Ganz korrekt ist dieser Vergleich aber nicht. Die DNA der 10 nm-Faser geht ja nicht durch die einzelnen Nucleosomen hindurch, sondern wickelt sich um die Histone des Core-Komplexes herum.
Das obige Bild täuscht außerdem in einer zweiten Hinsicht. Die "Perlenkette" der Nucleosomen ist keineswegs so regelmäßig aufgebaut, wie man es hier und in den meisten Fachbüchern sieht. Es gibt bestimmte Bereiche in der DNA, in denen die Nucleosomendichte sehr gering ist, und andere Bereiche mit einer höheren Nucleosomendichte, zum Beispiel im Centromer der Chromosomen. In Bereichen mit niedriger Nucleosomendichte können sich RNA-Polymerasen und Transkriptionsfaktoren leichter an die DNA-Doppelhelix anlagern, um die Transkription von Genen durchzuführen.
Stufe 3: 30-nm-Faser
Unter bestimmten Bedingungen bildet diese 10 nm-Faser eine Spirale mit einem Durchmesser von 30 nm, die als 30-nm-Faser oder 30-nm-Filament bezeichnet wird (engl.: 30-nm chromatin fiber). Das Histon H1 ist bei dieser Faserbildung beteiligt, wie Versuche ergeben haben. Entfernt man H1 experimentell, so löst sich die 30-nm-Faser auf, fügt man H1 hinzu, bildet sich die 30-nm-Faser wieder zurück [3]. Eine Windung dieser 30-nm-Faser besteht aus sechs Nucleosomen.
Durch die Bildung von 30-nm-Fasern wird die Länge der DNA insgesamt um den Faktor 40 reduziert [6].
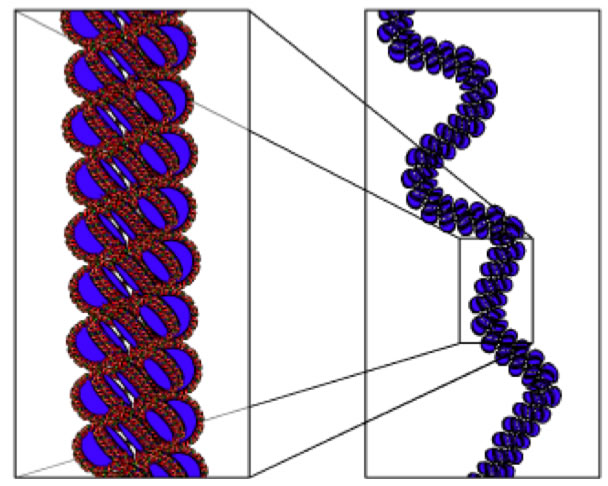
Eine grobe Darstellung der 30 nm-Faser
Quelle: Wikipedia, Artikel "Solenoid (DNA)", Autor: Richard Wheeler, Lizenz: Creative Commons Attribution-ShareAlike 4.0.
Es gibt allerdings auch noch andere Strukturen, je nach Salzgehalt und pH-Wert des Milieus. Eine Windung dieser Spirale besteht aus sechs Nucleosomen.
Nach neueren Erkenntnissen gibt es aber Zweifel an der alleinigen Existenz dieser Struktur der 30-nm-Faser [5]. Es wurden eine Reihe alternativer Strukturen gefunden bzw. vorgeschlagen, die bekannteste Alternativ-Struktur ist das Zick-Zack-Modell, das genau so wie die klassische Solenoid-Struktur elektronenmikroskopisch beobachtet worden ist.
Die DNA, die sich in einer solchen kompakten 30-nm-Solenoid- oder Zick-Zack-Struktur befindet, kann nicht transkribiert werden. Vor der Transkription müssen diese Strukturen aufgelöst werden.
In ihrem Open Access Artikel "The 10-nm chromatin fiber and its relationship to interphase chromosome organization" schlagen Jeffrey C. Hansen et al. sogar ein völlig anderes Modell vor. Ihrer Meinung nach sind die Nucleosomen viel "chaotischer" organisiert, zumindest in den Bereichen, in denen Gene transkribiert werden.
"In the new view, the default conformation of genomic chromatin is the 10-nm fiber..., and it is the 30-nm fiber that may exist only transiently, or for specific regulatory purposes such as terminal differentiation." [7]

In vitro conformational dynamics of the chromatin fiber
Autor: Jeffry C. Hansen et al., Lizenz: Creative Commons Attribution License 4.0 (CC BY-NC-ND).
Stufe 4: 300 nm-Faser
Bei der "Aufwicklung" der 30 nm-Faser zu einer zehnmal so dicken Struktur spielen die Histone keine Rolle mehr, jetzt kommen andere Proteine ins Spiel. Nach neueren Erkenntnissen bilden diese Proteine quasi das Grundgerüst eines Chromosoms, in das dann die 30 nm-Fasern "eingehängt" werden. Unter bestimmten Bedingungen kann man im Elektronenmikroskop als nächsthöhere Struktur Fasern mit 250 bis 300 nm Durchmesser erkennen.
Durch die Bildung von 300-nm-Fasern wird die Länge der DNA insgesamt um den Faktor 680 reduziert [6].
Stufe 5: 700 nm-Faser
Die 300 nm-Fasern wickeln sich dann nochmals zu Fasern von ca. 700 nm Durchmesser auf. Diese Fasern schließlich sind dann "aufgewickelt" zum eigentlichen Chromosom.
Durch die Bildung von 700-nm-Fasern wird die Länge der DNA insgesamt um den Faktor 12.000 reduziert [6].
Stufe 6: Chromosom
Damit wären wir am Ende der DNA-Kondensation.
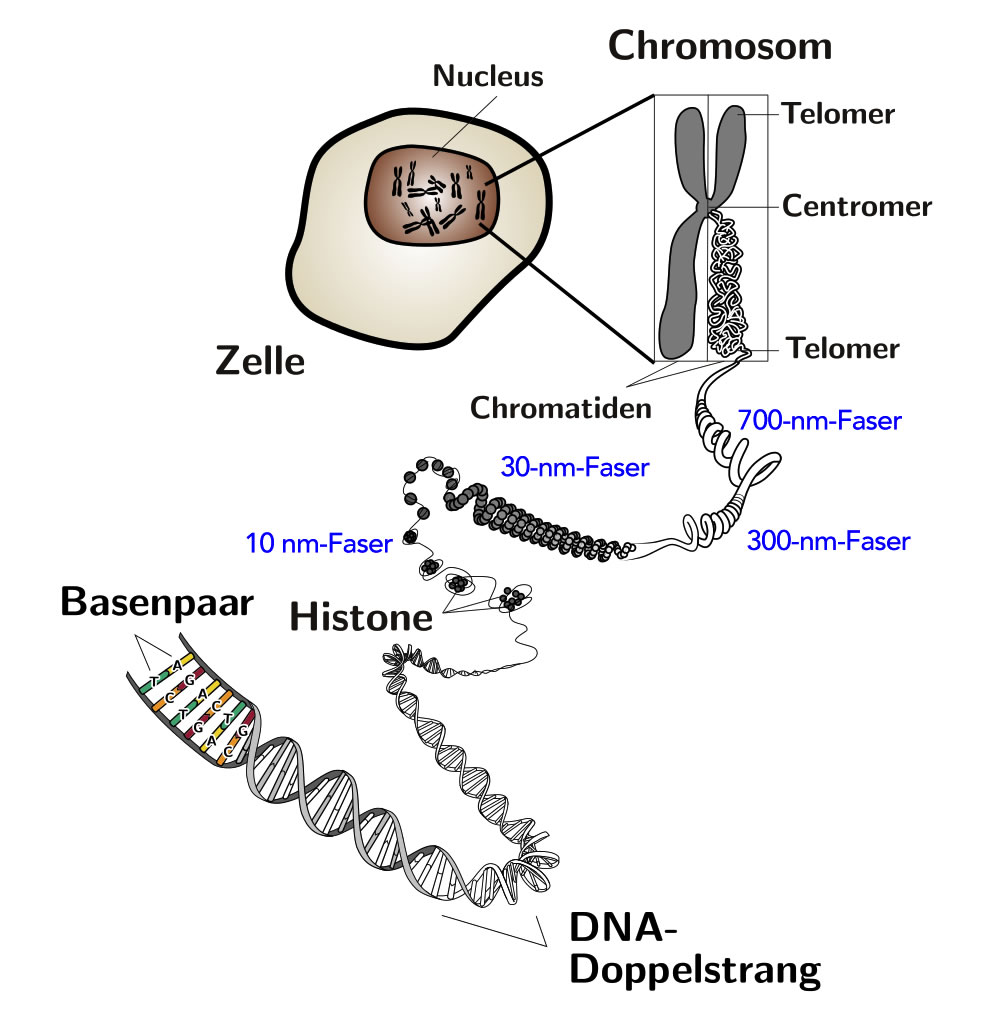
Typisches Lehrbuchbild des Chromosomenaufbaus
Quelle: commons.wikimedia.org, Autor: Phrood~commonswiki, Lizenz: public domain.
Hier noch einmal ein schönes Bild, wie man es aus den meisten Oberstufen-Lehrbüchern kennt. Ob ein Chromosom tatsächlich so aufgebaut ist, ist noch nicht endgültig geklärt. Allein über die genaue Struktur der 30-nm-Phaser gibt es kontroverse Ansichten, ist sie eher spiralig aufgebaut oder eher zick-zack-förmig oder vielleicht so, wie es Hansen et al. in [7] vorschlagen?
Quellen:
- Alberts et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.
- Knippers, Molekulare Genetik, Stuttgart 2006.
- Spektrum-Lexikon der Biologie, Artikel "Histon-Acetylierung".
- Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Spektrum-Lexikon der Biologie, Artikel "Chromatinorganisation".
- Hansen et al.: "The 10-nm chromatin fiber and its relationship to interphase chromosome organization", Biochem Soc Trans. 2018 Feb 19; 46(1): 67–76.