Abituraufgaben
Dieses Thema wird von folgenden NRW-Abituraufgaben behandelt:
- 2021 GK HT 3: Das Warmblood Fragile Foal Syndrome (WFFS)
- 2021 LK HT 3: Erbliche Netzhauterkrankung
Grundsätzliche Überlegungen
Bei der Translation müssen 20 bis 21 Aminosäuren in das wachsende Protein eingebaut werden. Die Information, welche Aminosäuren wann einzubauen ist, beziehen die Ribosomen aus der mRNA, die ja aus genau vier "Buchstaben" besteht: A, C, G und U.
Eine einfache mathematische Überlegung zeigt nun, dass "Worte" aus zwei dieser Buchstaben (z.B. GC) nur für die Codierung von 16 Aminosäuren reichen würden. Worte aus drei Buchstaben (z.B. GCA) reichen für die Verschlüsselung von 64 Aminosäuren, und Worte aus vier Buchstaben (z.B. GCAA) würden sogar für 256 Aminosäuren reichen.
In der Evolution setzen sich immer (oder fast immer) die ökonomisch günstigsten Varianten durch, und das wäre hier ein Code, der sich aus 3-Buchstaben-Worten zusammensetzt. Weniger geht nicht für 21 Aminosäuren, und mehr wäre unökonomisch.
Die Experimente von Nirenberg & Co.
Versuch 1
Das einfachste Experiment, mit dem man den genetischen Code aufklären kann, ist die in-vitro-Translation künstlicher mRNAs, die nur aus einer Sorte von Nucleotiden bestehen. MATTHAEI und NIRENBERG führten entsprechende Versuche 1961 durch.
Alles begann mit Escherichia coli-Bakterien. Diese wurden homogenisiert (aufgeschlossen) und dann zentrifugiert. So konnte man die schwereren Zellbestandteile von den leichteren (mRNA, tRNA, Ribosomen, Enzyme etc.) abtrennen. Die leichteren Komponenten überführte man dann in ein neues Reagenzglas. In vitro (im Glas) konnte man dann eine Peptidsynthese beobachten, es wurden neue Peptide hergestellt. Gab man radioaktiv markierte Aminosäuren in das Reagenzglas, so entstanden kurze Zeit später radioaktiv markierte Peptide. Das war der Beweis, dass tatsächlich eine in-vitro-Translation stattfand.
Dummerweise war bei der Zentrifugation und Abtrennung der leichteren Zellbestandteile auch jede Menge zelleigener mRNA mit in das Reagenzglas gekommen, deren Zusammensetzung natürlich völlig unbekannt war. Diese mRNA musste man erst mal loswerden. Dazu gaben Nierenberg und Matthaei Ribonucleasen in das Reagenzglas, welche die zelleigene mRNA zerstörten. Anschließend wurde ein großer Überschuss an künstlicher Poly-U-mRNA dazugegeben, und die Translation der künstlichen mRNA konnte theoretisch losgehen.
Wie fand man nun heraus, dass tatsächlich eine Translation stattfand, und wie bekam man heraus, welche Aminosäure(n) in das neue Peptid eingebaut wurde(n)?
Es gibt ja bekanntlich 20 verschiedene biogene Aminosäuren. In das Reagenzglas wurden nun Exemplare einer jeden Aminosäure gegeben, aber nur eine Aminosäure war dabei radioaktiv markiert. Beim ersten Durchgang hat man beispielsweise die Aminosäure Arginin markiert. Nach erfolgter Translation musste man nun untersuchen, ob das hergestellte Peptid radioaktiv war. War dies der Fall, dann wusste man, dass die Aminosäure Arginin eingebaut worden war.
In der Praxis verfuhr man folgendermaßen, um herauszubekommen, welche Aminosäure(n) eingebaut wurde(n). Man goss den Inhalt des Reagenzglases auf einen sehr feinen Filter. Alle Aminosäuren, die nicht in das Peptid eingebaut worden waren, konnten die kleinen Poren dieses Filters passieren. Das hergestellte Peptid jedoch nicht, es war schon zu groß für die Poren und bliebt auf dem Filter liegen.
Im Falle der Poly-U-mRNA und der radioaktiv markierten Aminosäure Arginin fand man auf dem Filter keine auffällig hohe Radioaktivität, nur geringe Spuren. Daraus konnte man schließen, dass Arginin auf gar keinen Fall in das Peptid eingebaut worden war. Der Code UUU stand also nicht für die Aminosäure Arginin.
Insgesamt musste man den Versuch jetzt 19 mal wiederholen - einmal für jede der 19 anderen Aminosäuren. Als man die Aminosäure Phenylalanin radioaktiv markierte, zeigt sich auf dem Filter eine hohe Radioaktivität. Das war der Beweis dafür, dass das Codon UUU für die Aminosäure Phenylalanin steht, denn das Peptid enthielt jetzt jede Menge dieser Aminosäure.
Bei den anderen 19 Aminosäuren fand man keine nennenswerte Radioaktivität auf dem Filter, das heißt, es wurde nur Arginin in das Peptid eingebaut. Damit hatte man also den Code für Arginin entdeckt - 20 Teilversuche waren dafür notwendig, und es gab noch 63 weitere Codons, die man noch nicht analysiert hatte.
Es stellt sich nun die Frage: Wie viele Codons kann man mithilfe solcher Experimente entschlüsseln? Die Antwort darauf lautet: Genau vier. Statt Poly-U kann man den Versuch mit Poly-A, Poly-G und Poly-C durchführen. Und so fand man heraus, dass AAA für Lysin steht, CCC für Prolin, UUU für Phenylalanin. Mit der Poly-G-mRNA funktionierte der Versuch allerdings nicht. Eine Poly-G-mRNA bildet nämlich eine Sekundärstruktur, die sich nicht an die Ribosomen anlagern kann. Damit waren schon einmal drei der 64 möglichen Codes für Aminosäuren "geknackt".
Eine sehr ausführliche Darstellung der Versuche von Nirenberg und Matthaei (englisch).
Versuch 2
Schauen wir uns nun das nächst komplizierte Experiment an, das ebenfalls Anfang der 60er Jahre durchgeführt wurde. Man stellte wieder künstliche mRNA her, war diesmal aber etwas abwechslungsreicher als bei den allerersten Versuchen. Betrachten wir zum Beispiel folgende künstliche mRNA:
U G U G U G U G U G U G U G U G U G
Wie viele Codons mögen in dieser mRNA enthalten sein? Da wir schon wissen, dass ein Codon immer aus drei Basen besteht, können wir uns leicht ausrechnen, dass die mRNA genau zwei Codons enthält, nämlich UGU und GUG:
U G U G U G U G U G U G U G U G U G
Da diese Poly-UG-RNA nur zwei verschiedene Codons enthält, sollte das gebildete Protein auch nur zwei verschiedene Aminosäuren enthalten, die sich regelmäßig abwechseln. Und tatsächlich, genau das kam bei diesem Experiment heraus. Es entstand folgendes Protein:
Cys - Val - Cys - Val - Cys - Val
Allerdings kann man hier noch nicht genau sagen, welcher Code für welche Aminosäure steht. UGU steht entweder für Cystein oder Valin, und GUG steht ebenfalls entweder für Cystein oder Valin. Mit anderen künstlichen mRNAs aus zwei sich regelmäßig abwechselnden Basen konnte man den Code weiterer Aminosäuren "knacken" - den ganzen genetischen Code konnte man auf diese Weise aber nicht aufklären.
Versuch 3
Das Grundprinzip dieses Versuchs wollen wir uns an einem sehr einfachen Beispiel klar machen. Angenommen, wir geben mRNA-Nucleotide A und G im Verhältnis 2 : 3 in ein Reagenzglas und lassen diese Nucleotide zu einer künstlichen mRNA aggregieren. Die Reihenfolge der zwei Basen ist dann rein zufällig, aber die Zusammensetzung der mRNA ist uns wohlbekannt: 2/5 A und 3/5 G bzw. 40% A und 60% G.
Die Wahrscheinlichkeit, in dieser mRNA ein AAA-Codon vorzufinden, beträgt genau 2/5 * 2/5 * 2/5 = 8/125 bzw. 6,4%. Die Wahrscheinlichkeit, ein AAG, ein AGA oder ein GAA-Codon zu finden, beträgt genau 2/5 * 2/5 * 3/5 = 12/125 bzw. 9,6%
Die folgende Tabelle zeigt die genauen Wahrscheinlichkeiten, mit der die einzelnen Codons auftreten:
| Codon | W(Codon) | AS |
| AAA | 2/5 * 2/5 * 2/5 = 8/125 | Lys |
| AAG | 2/5 * 2/5 * 3/5 = 12/125 | Lys |
| AGA | 2/5 * 3/5 * 2/5 = 12/125 | Arg |
| GAA | 3/5 * 2/5 * 2/5 = 12/125 | Glu |
| AGG | 2/5 * 3/5 * 3/5 = 18/125 | Arg |
| GAG | 3/5 * 2/5 * 3/5 = 18/125 | Glu |
| GGA | 3/5 * 3/5 * 2/5 = 18/125 | Gly |
| GGG | 3/5 * 3/5 * 3/5 = 27/125 | Gly |
Fassen wir die Wahrscheinlichkeiten zusammen, mit denen wir die vier Aminosäuren in dem Peptid vorfinden, das durch die in-vitro-Translation entsteht, wenn wir die richtigen Komponenten zufügen:
Lysin = 20/125, Arginin = 30/125, Glutamin = 30/125 und Glycin = 45/125. Zusammen sind das 125/125, die Rechnung geht also auf.
In den 60er Jahren konnte man natürlich noch nicht wissen, dass das Codon AGG für die Aminosäure Arginin steht, man konnte nur berechnen, dass das Codon AGG mit einer Wahrscheinlichkeit von 18/125 = 14,4% in der künstlichen mRNA vorkommt.
Die prozentuale Codon-Zusammensetzung der künstlichen mRNA war also bekannt. Und das durch Translation gebildete Peptid konnte schon recht gut analysiert werden. Man konnte also herausfinden, welche Aminosäure zu wie viel Prozent in diesem Peptid vorkommt.
Dann musste man nur noch die Ergebnisse dieser Peptid-Analyse mit den zuvor berechneten Codon-Wahrscheinlichkeiten vergleichen:
- Die Aminosäure Lysin kam zu 16% in dem Peptid vor,
- das Codon AA* (AAA + AAG) trat mit einer Wahrscheinlichkeit von 16% in der mRNA auf.
Aus solchen Vergleichen konnte man dann schlussfolgern, dass das Codon AA* für die Aminosäure Lysin zuständig ist.
Dummerweise gibt es 64 mögliche Codons, aber nur 20 bis 21 verschiedene Aminosäuren. Für manche Aminosäuren existieren also mehrere Codons, so auch in unserem obigen Beispiel. Lysin wird zum Beispiel sowohl durch AAA wie auch durch AAG codiert. Diese Redundanz des genetischen Codes machte anfangs natürlich Schwierigkeiten bei der Berechnung der Wahrscheinlichkeiten, aber durch geschicktes Experimentieren und mathematische Findigkeit konnten diese Probleme dann bald behoben werden. Einzelheiten erspare ich mir hier, die kenne ich selbst auch noch nicht. Wer Lust hat, kann da ja mal recherchieren und mir dann einen Gastbeitrag schicken, den ich hier veröffentlichen kann.
Versuch 4
Nirenberg und Leder synthetisierten 1964 mRNAs, die nur aus drei Nucleotiden bestanden. Man sollte nun denken: "Diese kurzen RNA-Moleküle können unmöglich die beiden Ribosomen-Untereinheiten an sich binden, dafür sind sie viel zu kurz!". Irrtum! Zum Glück für die Wissenschaftler bildeten sich tatsächlich solche Trinucleotid-Ribosomen-Komplexe.
Diese Komplexe konnten nur jeweils eine spezifische tRNA binden. An den Komplex aus der mRNA UGU und einem Ribosom band sich zum Beispiel die tRNA für die Aminosäure Cystein, also Cys-tRNA.
In der Theorie hört sich das gut an, doch wie bekam man in der Praxis heraus, dass sich Cys-tRNA an den UGU-Ribosomen-Komplex gebunden hatte und nicht beispielsweise Arg-tRNA?
Und auch hierfür hatten die Wissenschaftler um Nirenberg eine gute Idee: "Trinucleotid-Ribosomen-Komplexe wurden auf Membranfiltern mit einer bestimmten definierten Porengröße zurückgehalten, während die viel kleineren, freien, nicht ans Ribosom gebundenen tRNA-Moleküle ungehindert durch das Filter laufen." [3] Anschließend mussten die zurückgehaltenen Ribosomen-mRNA-tRNA-Komplexe analysiert werden. Da die Aminosäuren ähnlich wie bei den vorhergehenden Versuchen radioaktiv markiert waren ( immer nur eine Aminosäure pro Versuchsdurchgang), konnte man leicht bestimmen, welche Aminosäure auf dem Filter zurückblieb.
Innerhalb eines Jahres konnte so der komplette genetische Code entschlüsselt werden und als Codesonne oder Codetabelle in jedem Oberstufen-Lehrbuch wiedergegeben werden.
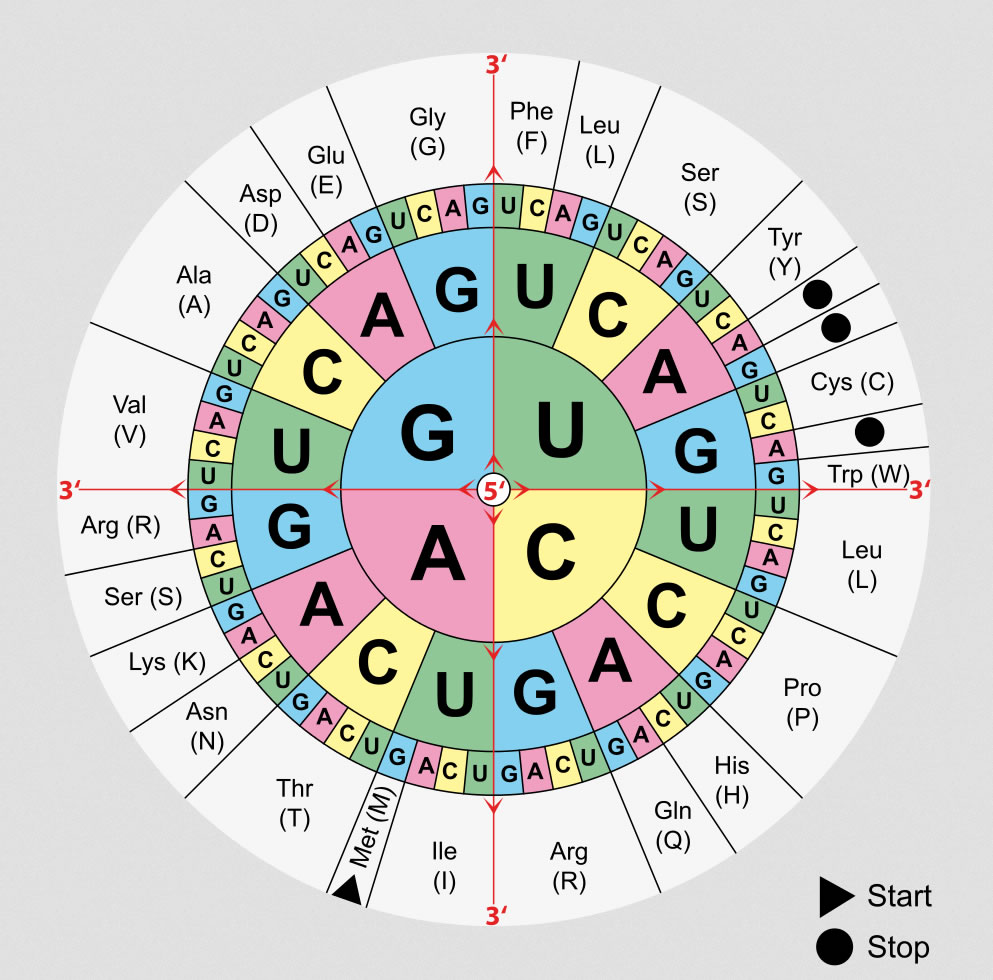
Die Codesonne
Quelle: Wikipedia, Artikel "Code-Sonne", Autor: Mouagip, Lizenz: public domain
Universalität des genetischen Codes
Alle Lebewesen der Erde haben den selben genetischen Code. Die Aminosäure Cystein wird beispielsweise stets durch die RNA-Tripletts UGU und UGC codiert. Man nimmt daher an, dass bereits die ersten prokaryotischen Zellen, die einen Proteinsynthese-Apparat besaßen, diesen Code "erfunden" haben. Die Universalität des genetischen Codes ist einer der vielen Belege für die Richtigkeit der modernen Evolutionstheorie.
Aber genau wie manche Fische oder Amphibien, die ständig in dunklen Höhlen leben, ihre Augen reduziert oder ganz verloren haben, gibt es auch ein paar Abweichungen und Ausnahmen beim genetischen Code in Folge evolutionärer Anpassungen.
Fallbeispiel Krebszellen [4]
Krebszellen werden vom menschlichen Immunsystem beispielsweise von bestimmten Signalmolekülen wie Interferon bekämpft. Allerdings wissen sich die Krebszellen zu wehren, sie stellen mehr von einem Enzym her, das die Aminosäure Tryptophan abbaut und damit auch das Interferon unschädlich macht. Allerdings wird die Trp-Konzentration in der Krebszelle ebenfalls verringert, quasi ein Kollateralschaden.
Wie man aber kürzlich herausgefunden hat, kompensieren die Tumorzellen diesen selbst verursachten Trp-Mangel durch einen genialen Schachzug: Die tRNA für die Aminosäure Tryptophan wird einfach mit Phenylalanin beladen. Diese Aminosäure hat eine ähnliche Struktur wie Tryptophan, und die Eigenschaften der hergestellten Proteine verändern sich nicht allzu stark, wenn statt Tryptophan das ähnliche Phenylalanin eingebaut wird.
Was jetzt aber für die Genetik interessant ist: Der genetische Code für Tryptophan hat sich jetzt verändert.
Quellen:
- Alberts, Bruce et al. Lehrbuch der Molekularen Zellbiologie, 5. Auflage, Weinheim 2021.
- Tiwari. 3 Kinds of Approaches by which the Genetic Code has been Cracked or Deciphered. Auf www.preservearticles.com.
- Knippers, molekulare Genetik, 1995
- Baranov, Atkins: "Krebszellen ändern ihren genetischen Code" in Spektrum der Wissenschaft 9/22, S. 28ff