Lernziele
Wenn Sie diese Seite durchgearbeitet haben, sollten Sie
- die Aufgaben der Transkription beschreiben können,
- den Unterschied zwischen Transkription und DNA-Replikation aufzeigen können,
- den Ablauf der Transkription in Einzelschritten erläutern können,
- den Aufbau eines Operons zeichnen und erläutern können,
- dabei die Funktionen von Promotor und Operator näher erklären können,
- Die Rollen von Promotor, Operator, Strukturgenen und RNA-Polymerase beim Transkriptionsvorgang erläutern können.
Zusammenfassung
Bei der Transkription wird eine mRNA-Kopie eines Gens der DNA erstellt, das dafür notwendige Enzym ist die RNA-Polymerase. Dieses Enzym bindet zunächst an den Promotor des betreffenden Gens, und dann wird einer der beiden DNA-Stränge, der codierende oder codogene Strang, in einen komplementären mRNA-Strang übersetzt.
1. Aufgabe der Transkription
Die Aufgabe der Transkription besteht in der Bereitstellung einer Arbeitskopie eines DNA-Abschnitts, zum Beispiel eines Strukturgens. Diese Arbeitskopie in Form einer mRNA wird dann von den Ribosomen in ein Protein übersetzt.
Aber auch die ribosomale RNA - die rRNA - die ein wichtiges Strukturelement der Ribosomen darstellt, muss durch Transkription der entsprechenden DNA-Abschnitte bereitgestellt werden. Und auch die tRNA-Moleküle einer Zelle entstehen nicht einfach so, sondern durch Transkription entsprechender Gene auf der DNA.
Vieles ist noch ungeklärt, was die Transkription der DNA angeht. Werden alle Abschnitte der DNA transkribiert? Was ist mit der so genannten "Schrott-DNA", die sich zwischen den Genen befindet und die sogar den größten Teil der DNA ausmacht?
Ein Teil dieser "Schrott-DNA" wird tatsächlich transkribiert, wie man bereits vor 20 Jahren festgestellt hat, aber welche Rolle die so gebildeten RNA-Moleküle spielen, ist weitgehend noch ungeklärt. Man nimmt an, dass diese RNA u.a. eine Rolle bei der Genregulation spielt.
2. Unterschied Transkription - Replikation
Die Transkription ist im Grunde eine abgewandelte Replikation. Auch evolutionsgeschichtlich nimmt man an, dass die Transkription aus der Replikation hervorgegangen ist. Allerdings gibt es einige Unterschiede zwischen der Replikation der DNA und der Transkription (und diese Unterschiede werden in Biologie-Klausuren oder Abitur-Prüfungen manchmal abgefragt):
Erstens
- Es wird nicht die gesamte DNA einer Zelle transkribiert, sondern nur ein kleiner Teil, nämlich ein Gen oder ein Operon, also eine kleine, zusammengehörige Gruppe von Genen.
Unter einem Gen verstehen wir einen DNA-Abschnitt, der für die Synthese eines Proteins, einer rRNA oder einer tRNA verantwortlich ist. Der Begriff Operon ist weiter unten erklärt.
Zweitens
- Bei der Replikation werden beide Stränge der Doppelhelix kopiert, es entstehen zwei neue DNA-Stränge. Bei der Transkription wird nur von einem der beiden DNA-Stränge ein RNA-Strang angefertigt.
Drittens
- Bei der Replikation entsteht neue DNA. Die Abschrift, die bei der Transkription entsteht, ist keine DNA, sondern RNA. Die RNA (Ribonucleinsäure) unterscheidet sich von der DNA (Desoxyribonucleinsäure) in drei Punkten:
a) Die Zucker-Einheiten enthalten ein Sauerstoffatom mehr,
b) statt der Base Thymin kommt die Base Uracil in der RNA vor,
c) die RNA ist grundsätzlich nur einsträngig, kann allerdings durch komplementäre Paarung einzelner Abschnitt auch zweisträngige Bereiche aufweisen (Haarnadelschleifen).
Viertens
- Bei der Replikation verbleibt die DNA-Kopie im Zellkern bzw. bei Prokaryoten (die ja keinen Zellkern haben) in der Nähe der alten DNA.
Bei der Transkription dagegen wandert die neu synthetisierte RNA in das Zellplasma, wo sie sich mit Ribosomen zusammenlagert.
3. Ablauf der Transkription bei Bakterien
Das wichtigste Enzym bei der Transkription ist die RNA-Polymerase, die von der Funktionsweise durchaus mit der DNA-Polymerase gleichzusetzen ist. Wie wir aber gerade gesehen haben, wird bei der Transkription nicht die gesamte DNA kopiert, sondern nur der Teil, der für die Synthese eines Proteins (oder einer Gruppe von Proteinen) verantwortlich ist, ein so genanntes Gen (oder Operon). Dazu muss die RNA-Polymerase den Anfang dieses Genes bzw. Operons finden. Eine menschliche Zelle hat 2 x 46 DNA-Stränge im Zellkern (46 Chromosomen mit je zwei Chromatiden), wobei jeder ein paar Millionen Basenpaare lang ist. Wie soll die RNA-Polymerase da den Anfang eines Gens finden?
3.1 Aufbau eines bakteriellen Operons
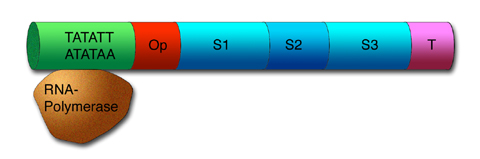
Ein prokaryotisches Operon
Autor: Ulrich Helmich, Lizenz: siehe Seitenende
Ein Operon ist sozusagen die kleinste funktionelle Einheit der DNA. Man könnte auch sagen: Ein Operon ist eine Gruppe zusammengehöriger Gene mit einem gemeinsamen Ein/Ausschalter.
Promotor
Am Anfang eines jeden Operons befindet sich der Promotor. Das ist ein kurzer DNA-Abschnitt, der durch eine bestimmte Basenabfolge gekennzeichnet ist, in der die Buchstaben T und A besonders häufig vorkommen. Der Promotor ist die Andockstelle für die RNA-Polymerase.
Ein Grund für das gehäufte Vorkommen der Basen T und A ist sicherlich, dass sich zwischen T- und A-Basen nur zwei Wasserstoffbrücken ausbilden. T- und A-reiche DNA-Regionen können also besonders leicht aufgetrennt werden, und das ist ja auch der erste wichtige Schritt bei der Transkription der DNA. Am Promotor kann die DNA-Doppelhelix also wegen der vielen T- und A-Basen besonders leicht in Einzelstränge gespalten werden (Spektrum der Wissenschaft, März 2014, Seite 20).
Auf dieser Lexikonseite erfahren Sie mehr über Promotoren.
Operator
Als nächstes kommt bei einem typischen bakteriellen Operon die Operator-Region. An diese DNA-Region kann sich ein spezifisches Repressor-Protein andocken. Sitzt ein solches Repressor-Protein am Operator, so ist der Weg für die RNA-Polymerase blockiert und eine Transkription ist nicht möglich. Dies ist eine effektive Methode, mit der eine Bakterienzelle die Transkription von Genen verhindern kann, die gerade nicht benötigt werden.
Strukturgene
An den Operator schließen sich die "richtigen" Gene an, die für die Proteine verantwortlich sind. Solche Gene bezeichnet man auch als Strukturgene. Das bekannteste bakterielle Operon, das lac-Operon, hat beispielsweise drei Strukturgene.
Terminator
Genau so wie der Anfang eines Genes besonders gekennzeichnet werden ist, muss auch das Ende eines Transkriptionsabschnittes markiert werden. Eine solche spezifische Basenabfolge bezeichnet man auch als Terminator.
3.2 Pro- und Eukaryoten
Diese Beschreibung des Aufbau eines Operons bezieht sich auf die Verhältnisse bei Prokaryoten, speziell Bakterien. Eukaryotische Gene bzw. Operons sind wesentlich komplexer aufgebaut; die aktivierenden oder hemmenden DNA-Sequenzen können über verschiedene Stellen der DNA verteilt sein. Auf die Transkription bei Eukaryoten wird im nächsten Abschnitt näher eingegangen.
4. Der Transkriptionsvorgang
Die RNA-Polymerase diffundiert im Kernplasma (bei Prokaryoten im Zellplasma) herum und kommt irgendwann mit einem DNA-Strang in Kontakt. Das Enzym setzt sich dann auf die DNA-Doppelhelix wie eine Lokomotive auf einen Schienenstrang. Es gleitet jetzt auf der DNA entlang. Sobald die RNA-Polymerase auf eine Promotor-Sequenz stößt, entsteht eine stabilere Bindung.
Wenn der Operator, der sich meistens direkt hinter dem Promotor befindet, nicht durch ein Repressor-Protein blockiert ist, beginnt die RNA-Polymerase mit der Transkription. Die Basen der Strukturgene werden in mRNA übersetzt. Bei Prokaryoten setzen sich sofort Ribosomen an die neu entstandene mRNA und beginnen zeitgleich mit der Translation. Bei Eukaryoten geht das nicht so schnell. Die mRNA muss nämlich zuerst den Zellkern verlassen, vorher wird sie allerdings noch etwas bearbeitet (RNA-Spleißen). Erst dann können sich Ribosomen an die mRNA setzen und mit der Translation beginnen.
Wenn die RNA-Polymerase am Terminator ankommt, stoppt die Transkription, und das Enzym verlässt die Doppelhelix. Die Polymerase diffundiert jetzt wieder ziellos im Kernplasma herum, bis sie erneut zufällig in Kontakt mit der DNA kommt.
4.1 Der Transkriptionsvorgang - näher betrachtet
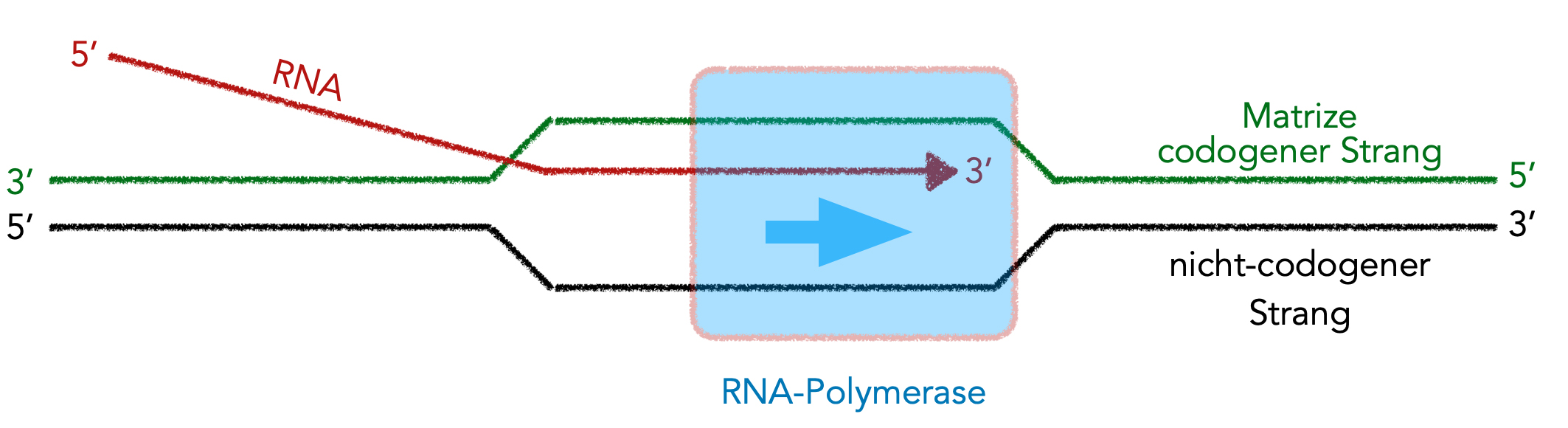
Der Transkriptionsvorgang schematisch dargestellt
Autor: Ulrich Helmich 2022, Lizenz: siehe Seitenende
Zunächst wird die DNA-Doppelhelix über eine Länge von mehreren Basenpaaren entwunden. Eine der beiden Einzelstränge ist dann die Matrize (Kopiervorlage) für die RNA; dieser Einzelstrang wird als codogener Strang oder auch als codierender Strang bezeichnet. Der andere DNA-Strang, der nicht kopiert wird, heißt nicht-codogener Strang oder nicht-codierender Strang.
Abiturienten aufgepasst!
In fast jeder Genetik-Klausur im Biologie-Abitur muss man eine kleine DNA-Sequenz in eine mRNA übersetzen. Sie müssen dabei ganz genau darauf achten, was in dem vorgegebenem Material steht. Wird die DNA-Sequenz des codogenen Strangs angegeben oder des nicht-codogenen Strangs?
Wenn die DNA-Sequenz des codogenen Strangs angegeben wird, müssen Sie die mRNA komplementär gestalten. Aus ACCTGCT wird dann beispielsweise UGGACGA. Wird aber die Sequenz des nicht-codogenen Strangs angegeben, brauchen Sie die DNA-Basen einfach nur abzuschreiben. Allerdings müssen Sie jedes T durch ein U ersetzen. Aus ACCTGCT auf dem nicht-codierenden Strang wird dann beispielsweise ACCUCGU.
An das wachsende RNA-Molekül werden jetzt von der RNA-Polymerase nach und nach die RNA-Nucleotide angehängt, und zwar an das 3'-OH-Ende der RNA.
Bei der Transkription werden allerdings keine Nucleotide "angeliefert", sondern Nucleosidtriphosphate, das sind quasi Nucleotide mit drei Phosphatgruppen.
Der Vorgang der Elongation (Verlängerung der mRNA) ist sehr energieaufwändig, und die RNA-Nucleosidtriphosphate bringen diese Energie selbst mit, in Form von zwei Phosphatgruppen, die dann abgespalten werden (ähnlich wie bei der Spaltung von ATP in AMP und 2 Phosphatreste).
Aufgepasst: Nucleoside, Nucleotide und Nucleosidtriphosphate
Da kann einem schon schwindelig werden bei diesen vielen Fachbegriffen. Also hier noch einmal ganz klar:
Nucleosid = Ribose + eine der vier Basen Adenin, Cytosin, Guanin oder Uracil.
Nucleotid = Nucleosid + Phosphatgruppe = Nucleosidmonophosphat
Nucleosidtriphosphat = Nucleosid + 3 Phosphatgruppen
Die RNA-Polymerase bewegt sich bei diesem Prozesse stetig weiter, und zwar in 3' → 5'-Richtung, wenn man den codogenen Strang als Bezugssystem wählt.
Auf dieser Lexikonseite erfahren Sie viele Details über pro- und eukaryotische RNA-Polymerasen, wie sie aufgebaut sind, welche Typen es gibt, und wie sie mit der DNA interagieren.
Auf dieser Vertiefungsseite erfahren Sie viele Details über die prokaryotische Transkription. Die Initiationsphase, die Elongationsphase und die Terminationsphase werden ausführlich beschrieben. Für Schüler(innen) der Sek. II ist diese Seite vielleicht schon zu anspruchsvoll, aber Sie können ja mal gerne rein schauen, vor allem, wenn Sie im Leistungskurs Biologie sind und schon überlegen, ob Sie vielleicht mal Biologie studieren sollten.