Abituraufgaben
Dieses Thema wird von folgenden NRW-Abituraufgaben behandelt:
- 2021 LK HT 3: Erbliche Netzhauterkrankung
Die Proteinsynthese der Eukaryoten unterscheidet sich in mehreren Punkten von der Proteinsynthese der Prokaryoten. Einige dieser Unterschiede wollen wir hier kurz auflisten.
Prokaryotische DNA
Die DNA der Prokaryoten liegt normalerweise "nackt" in der Zelle vor, weder gibt es eine Kernhülle, die sich schützend und abgrenzend um das Erbgut legt, noch gibt es nennenswerte Mengen von Proteinen, die der DNA eine übergeordnete Struktur verleihen, wie dies bei Eukaryoten der Fall ist (Histone, Chromosomen).
Eukaryotische DNA
Die DNA der Eukaryoten ist durch Histone und andere Proteine viel komplexer organisiert als die Prokaryoten-DNA, was es der RNA-Polymerase viel schwerer macht, die Transkription überhaupt durchzuführen. Wie soll sie an die eigentliche Erbinformation herankommen, wenn überall Proteine sind?
Zweitens ist das Erbgut der Eukaryoten durch eine Kernhülle vom Cytoplasma getrennt. Das wäre ja auch nicht weiter schlimm, würden sich nicht ausgerechnet die Ribosomen, also die Orte, an denen die Translation stattfindet, im Cytoplasma befinden. Also muss die mRNA, die sich während der Transkription bildet, aus dem Zellkern durch die Kernhülle in das Cytoplasma gelangen.
Prokaryotische Operons
Die Transkriptionseinheiten der Bakterien, Cyanobakterien (Blaualgen) und anderer Prokaryoten bestehen aus einem Promotor, einem Operator, einem oder mehreren Strukturgenen und schließlich einem Terminator. Spezifische Repressorproteine, die sich an den Operator setzen und so die Transkription verhindern können, werden durch ein Regulatorgen codiert. All diese Strukturelemente zusammen bilden dann ein so genanntes Operon.
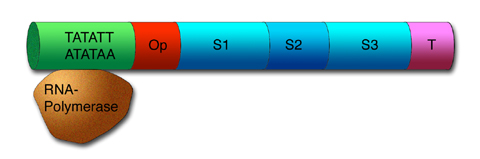
Ein prokaryotisches Operon - das Regulatorgen ist nicht eingezeichnet, es befindet sich irgendwo anders auf der DNA
Eukaryotische Operons
Ein eukaryotisches Operon ist im Prinzip ähnlich aufgebaut wie ein prokaryotisches, allerdings unterscheidet es sich in zwei Punkten. Erstens enthält ein eukaryotisches Operon stets nur ein Strukturgen und nicht mehrere. Und zweitens ist die Regulation eines eukaryotischen Operons wesentlich komplexer als die eines prokaryotischen. Bei einem prokaryotischen Operon gibt es einen spezifischen Repressor, der die RNA-Polymerase blockiert, wenn er am Operator sitzt. Bei manchen prokaryotischen Operons gibt es zusätzlich so genannte Aktivatoren, das sind Proteine, die in der Lage sind, die Geschwindigkeit der RNA-Polymerase zu erhöhen.
Statt dieser Aktivatoren und Repressoren gibt es in eukaryotischen Zellen jede Menge verschiedener Transkriptionsfaktoren, von denen manche fördernd (Enhancer), andere hemmend (Silencer) auf die Transkriptionsrate einwirken, teils auf sehr komplizierte Weise, die hier zu erläutern zu weit führen würde.
Eukaryotische Gene
Nicht nur die Operons unterscheiden sich in ihrem Aufbau, sondern auch die Gene selbst. Ein Prokaryoten-Gen besteht von vorne bis hinten aus codierenden Sequenzen. Ein Eukaryoten-Gen dagegen ist von nicht-codierenden Sequenzen durchsetzt, so genannten Introns(für "invervening sequences"). Die codierenden Sequenzen werden entsprechend als Exons (für "expressed sequences") bezeichnet.
Introns
Warum das so ist, weiß im Grunde keiner so richtig, es gibt aber viele Hypothesen. Zum Beispiel nehmen einige Biologen an, dass sich früher einmal Viren in das Genom der Eukaryoten integriert haben. Normalerweise erwacht so ein Provirus nach einiger Zeit zum Leben und infiziert dann die Zelle. Es gibt aber auch Fälle, wo sich ein Provirus über Jahre unauffällig verhält und einfach im Genom der Wirtszelle (irgendwo in die DNA eingebaut) verbleibt. Wenn sich die Wirtszelle teilt, wird auch ihre DNA repliziert, und mit der wirtseigenen DNA wird auch die eingebaute Viren-DNA repliziert. Introns könnten nun solche Proviren sein, die sich seit Millionen von Jahren in unserer DNA gehalten haben. Einige Biologen nehmen sogar an, dass uns solche DNA-Abschnitte vorteilhafte Eigenschaften verleihen.
Auch ein horizontaler Gentransfer zwischen verschiedenen Arten ist auf diese Weise möglich: Wenn sich ein Provirus aus der DNA löst und dabei benachbarte Gene "mitnimmt" und dann ein Individuum einer anderen Art infiziert, so verfrachtet er diese Fremdgene in das artfremde Individuum, wo sie sich vielleicht in dessen DNA integrieren.
Transkription eukaryotischer Gene
Im Grunde läuft die Transkription eines eukaryotischen Gens genau so ab wie die eines prokaryotischen Gens. Die RNA-Polymerase setzt sich an den Promotor, und wenn der Weg frei ist, wird das Gen komplett in mRNA umgeschrieben. Dabei werden sowohl die Exons wie auch die Introns transkribiert. Das Produkt dieser Transkription, die so genannte prä-mRNA, enthält also Exons und Introns.
Analysiert man aber die mRNA, wie sie aus dem Zellkern der Eukaryoten-Zelle herauskommt, stellt man fest, dass die Introns verschwunden sind. Die endgültige mRNA besteht nur noch aus den Exons.
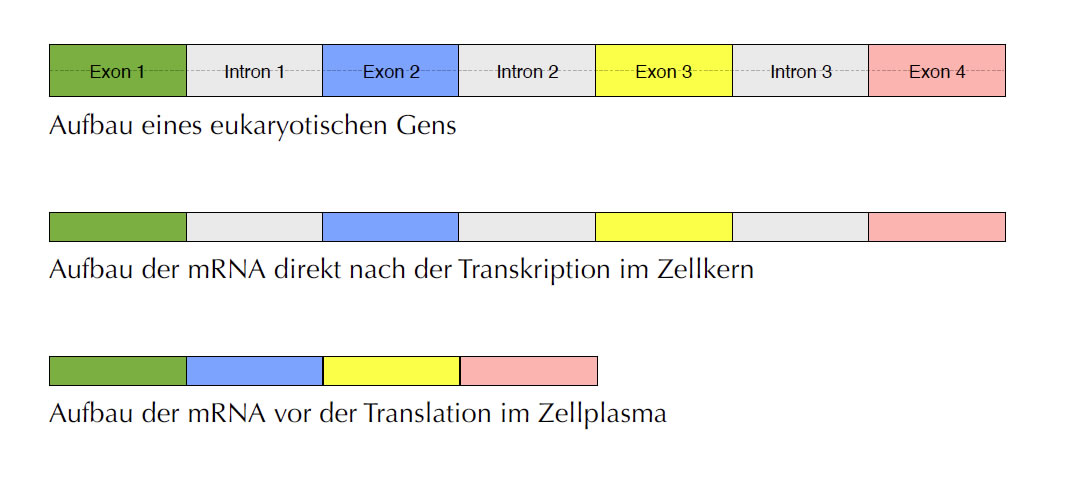
Prozessierung der prä-mRNA
Auf dem Bild sehen wir die DNA eines Gens, die aus vier Exons und drei Introns besteht. Bei der Transkription wird eine einsträngige Abschrift der gesamten DNA des Gens angefertigt, die prä-mRNA des Gens enthält daher alle Exons, aber auch alle Introns. Die mRNA des gleichen Gens, wenn sie im Cytoplasma ankommt, besteht aber nur noch aus den vier Exons. Also müssen die drei Introns irgendwo auf dem Weg vom Zellkern zum Cytoplasma verloren gegangen sein.
Spleißen
Weitere Nachforschungen haben dann ergeben, dass es im Zellkern besondere Enzyme gibt, die in der Lage sind, die Introns aus der prä-mRNA heraus zu schneiden. Diesen Vorgang des Herausschneidens von Introns nennt man im Englischen splicing und im Deutschen Spleißen. Die Enzyme, die dieses Spleißen bewerkstelligen, bezeichnet man dann als Spleißenzyme.
Ein anderer Begriff für die Aufarbeitung der prä-mRNA im Zellkern ist das RNA-Processing oder RNA-Prozessieren. Allerdings ist dieser Begriff etwas weiter gefasst als der des Spleißens. RNA-Processing bezeichnet alle Veränderungen, die an der prä-mRNA im Zellkern vorgenommen werden, während das Spleißen lediglich die Entfernung der Introns bedeutet.
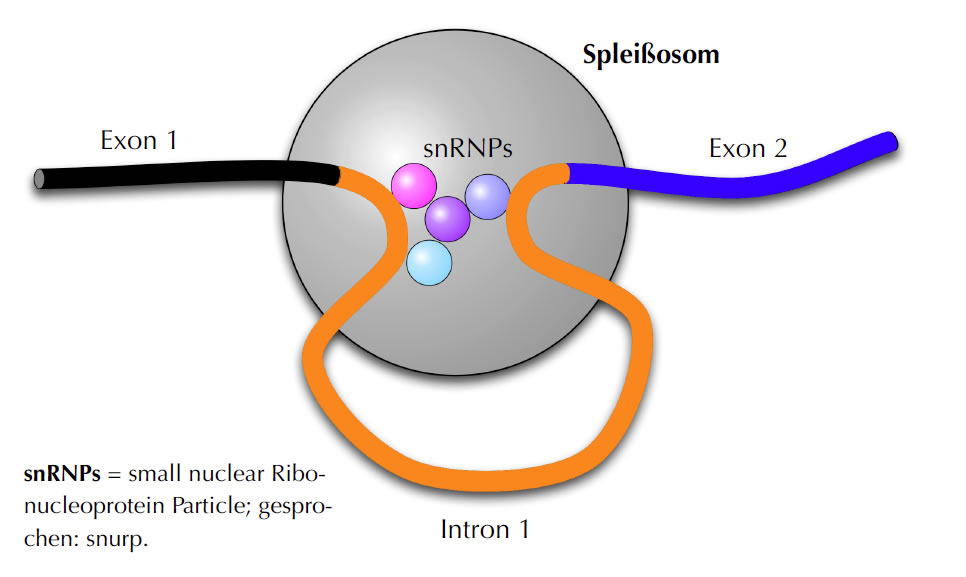
Entfernung eines Introns durch ein Spleißosom
Dieses Bild zeigt eine stark vereinfachte Vorstellung, wie das Spleißen funktionieren könnte. Mehrere Spleiß-Enzyme sowie einige snRNPs (das sind kleine Moleküle, die sich aus RNA und Proteinen zusammensetzen, ähnlich wie Ribosomen, aber viel kleiner) finden sich zu einem größeren Aggregat zusammen, das als Spleißosom (engl.: spliceosome) bezeichnet wird. Die snRNPs sorgen dafür, dass sich das Intron zu einer Art Haarnadelschleife faltet. Anschließend schneiden diese Enzyme das Intron aus der mRNA heraus und verknüpfen das 3'-Ende des ersten Exons mit dem 5'-Ende des zweiten Exons.

Das herausgeschnittene Intron und die beiden zusammengefügten Exons
Das war so ungefähr das Wichtigste zur Proteinsynthese bei Eukaryoten, was man als Grundkurs-Schüler wissen sollte. Wenn ich noch etwas Zeit habe, werde ich noch eine Vertiefungsseite für Leistungskurs-Schüler und Studenten anlegen. Mal sehen. Vielleicht schickt mir ja auch mal einer von meinen Lesern eine mit "Sehr gut" bewertete Facharbeit zum Thema, die ich dann hier gerne als Gastbeitrag veröffentliche.