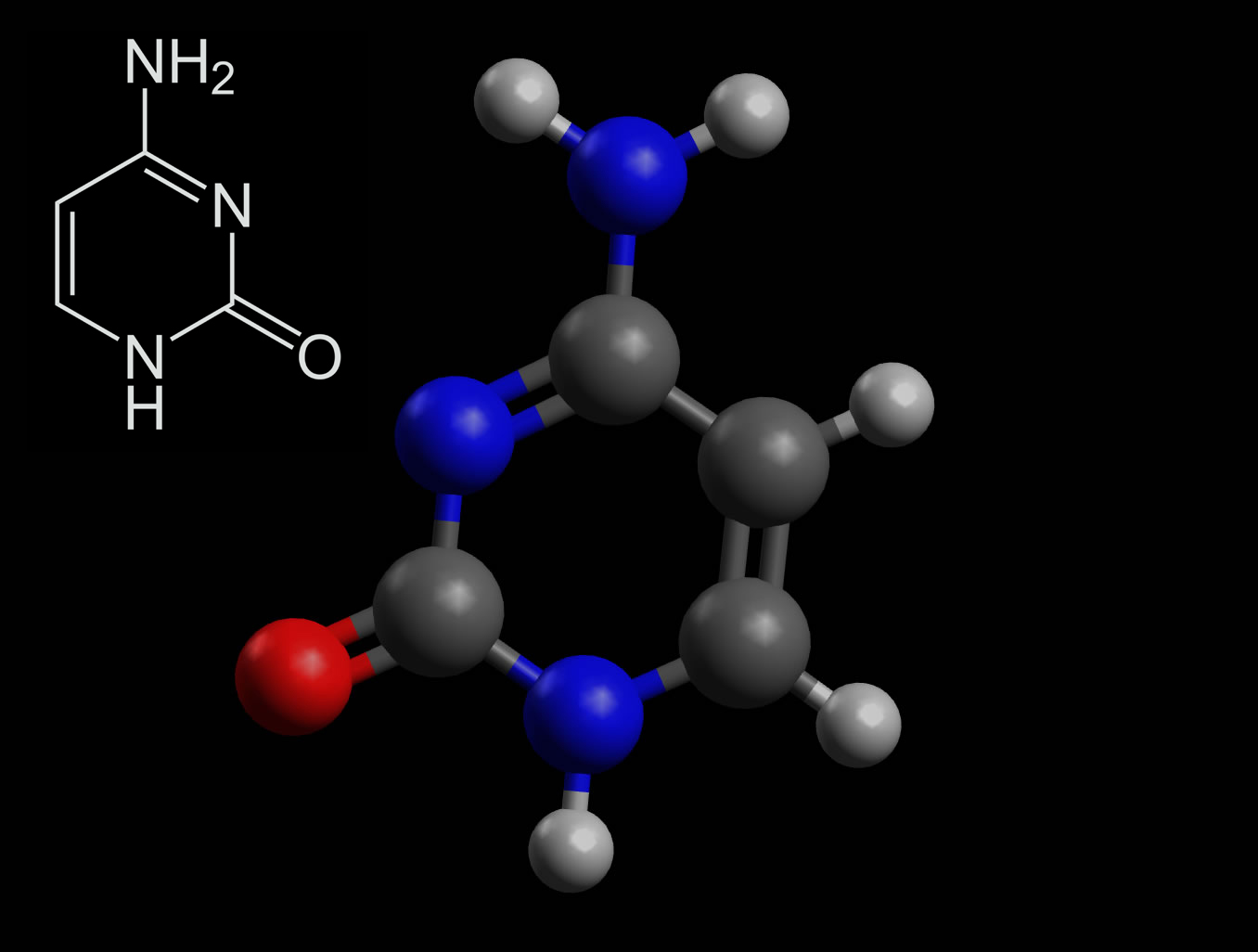
Das Cytosin-Molekül
Autor: Ulrich Helmich 2024, Lizenz: Public domain
Cytosin ist eine der vier DNA- bzw. RNA-Basen (neben Adenin, Guanin, Thymin und Uracil). Es handelt sich um eine fünfgliedrige heterozyklische Verbindung mit einem Pyrimidin-Grundgerüst.
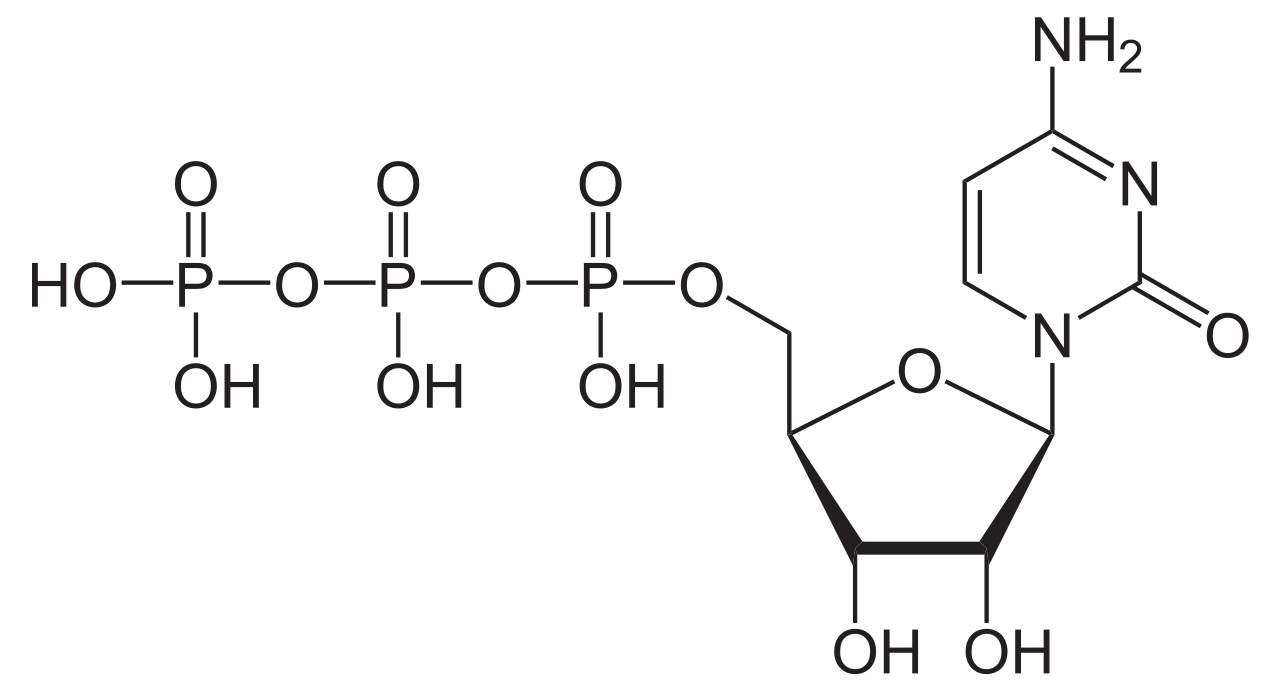
Cytidintriphosphat
NEUROtiker, Public domain, via Wikimedia Commons
Wenn die Base Cytosin in eine wachsende DNA- oder RNA-Kette eingebaut werden soll, wird es in Form von Cytidintriphosphat CTP angeliefert. Zwei der Phosphatgruppen werden als PPi abgespalten, und das Cytosin-Nucleotid wird dann in die DNA bzw. RNA eingebaut.
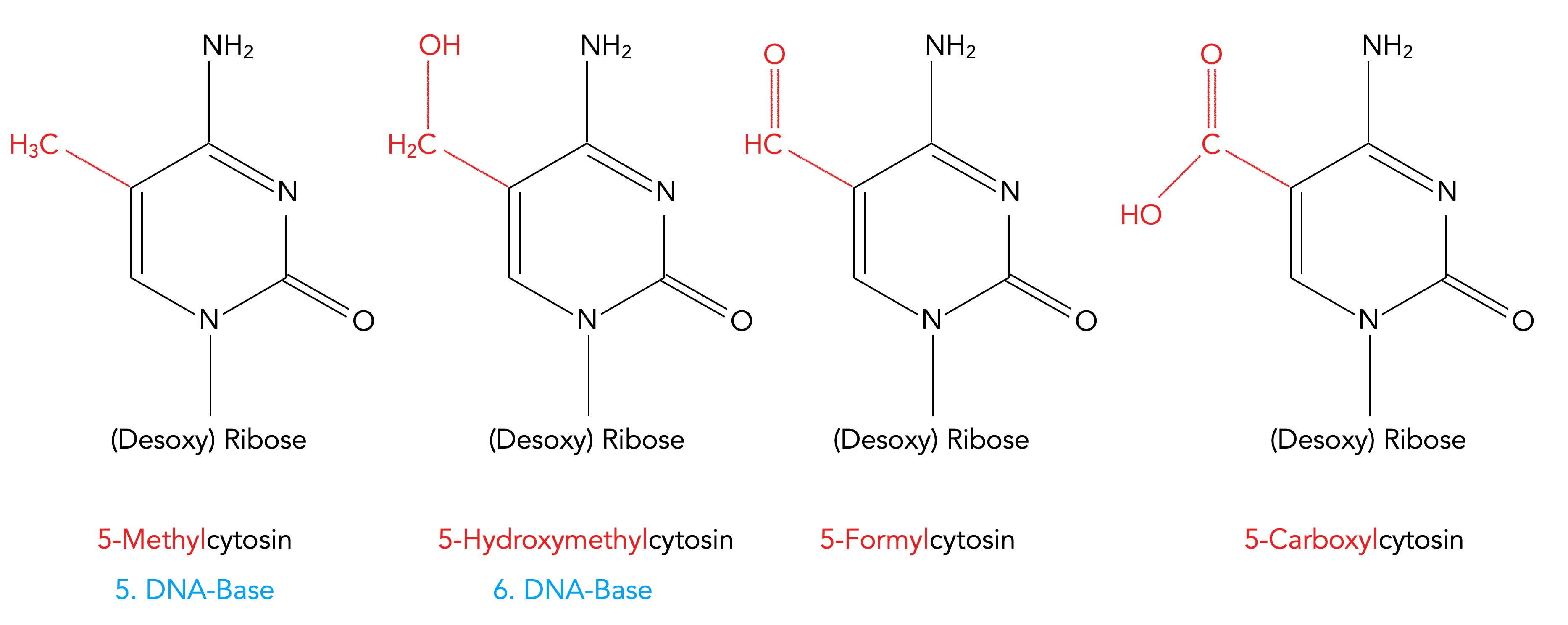
Modifizierte Cytosin-Basen
Neben der eigentlichen DNA/RNA-Base Cytosin findet man in der DNA oder RNA von Säugetieren chemisch modifizierte Cytosin-Bausteine.
Hier sehen wir die bekanntesten, in Säugetier-DNA gefundenen Cytosin-Modifikationen.
5-Methylcytosin
Diese modifizierte Base ist chemisch recht instabil und kann leicht zu Thymin umgewandelt werden:
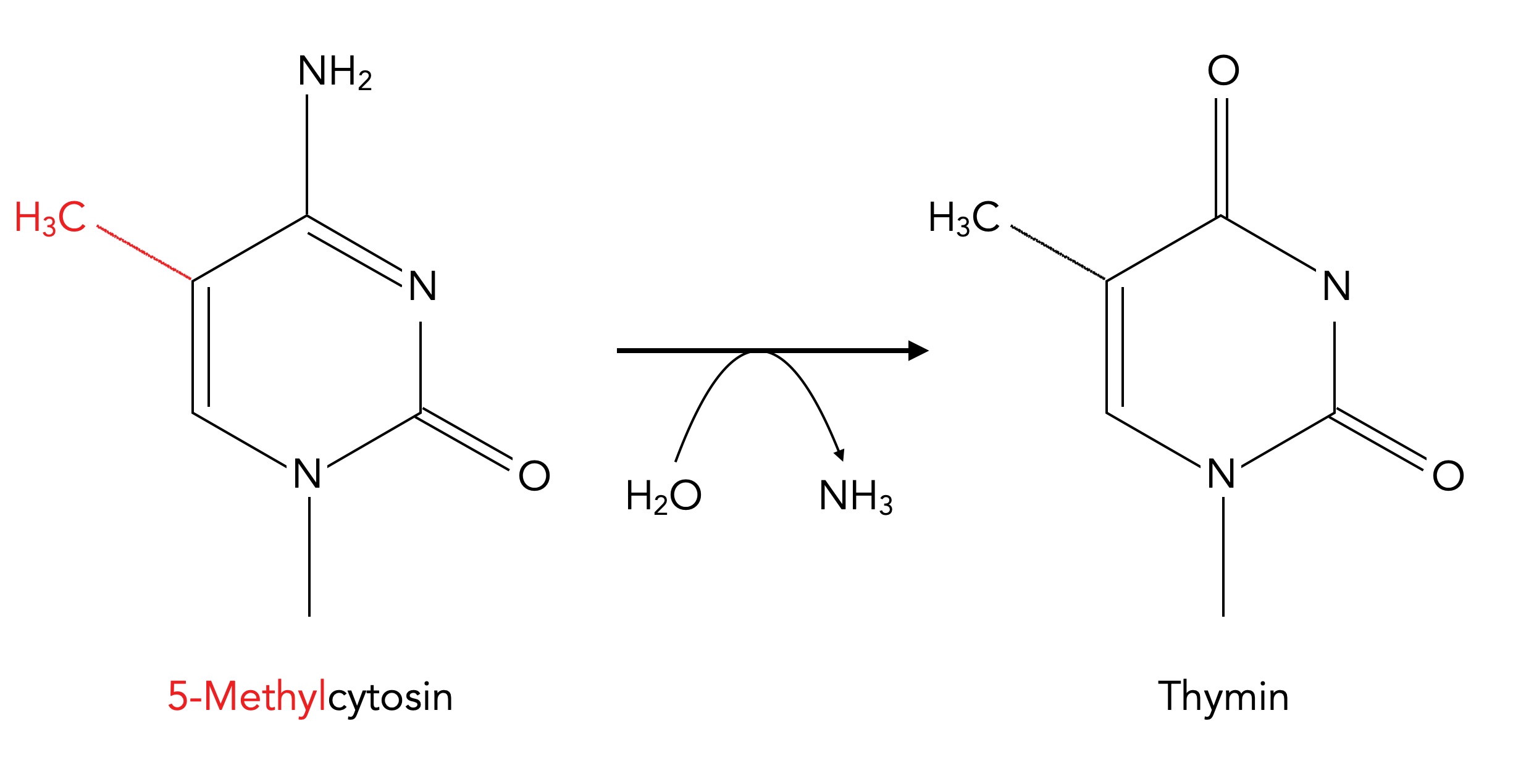
Desaminierung von 5-Methylcytosin zu Thymin
Autor: Ulrich Helmich 05/2024, Lizenz: Public domain
Wenn dies in einer lebenden Zelle passiert, so liegt eine Punktmutation vor. Bei der nächsten Replikationsrunde wird dann in dem neuen DNA-Strang nicht mehr Guanin (als Partner von Cytosin) eingebaut, sondern Adenin, der Partner von Thymin.
Wenn Sie mehr über diese Verbindung wissen möchten, die oft auch als fünfte DNA-Base bezeichnet wird, sind Sie auf dieser Wikipedia-Seite genau richtig.
5-Hydroxymethylcytosin
Dieses modifizierte Cytosin kommt sowohl in der DNA wie auch in der RNA von Säugetieren vor. 5-Hydroxymethylcytosin war die zweite modifizierte Cytosin-Base, die überhaupt entdeckt wurde. Daher bezeichnet man diese Base oft als sechste DNA-Base.
Genau wie 5-Methylcytosin wird auch 5-Hydroxymethylcytosin postreplikativ gebildet, also erst nach erfolgter Replikation der DNA.
Man nimmt an, dass beide modifizierten Basen, also sowohl 5-Methylcytosin wie auch 5-Hydroxymethylcytosin, eine wichtige Rolle in der Epigenetik spielen, also bei der umweltbedingten Modifikation der genetischen Information (DNA-Methylierung).
"2009 fanden zwei Forschergruppen heraus, dass 5-Hydroxymethylcytosin auch ein Bestandteil der DNA von Säugetieren ist. Inzwischen ist bekannt, dass praktisch jede Säugetierzelle 5-Hydroxymethylcytosin enthält und dass die größten Mengen im Zentralnervensystem vorkommen.
Die Menge an 5-Hydroxymethylcytosin nimmt während der Entwicklung zu, scheint aber im Erwachsenenalter stabil zu sein." [1]
Auch in der DNA von Bacteriophagen (Viren, die ausschließlich bestimmte Bakterien befallen) hat man 5-Hydroxymethylcytosin entdeckt. Dort scheint diese modifizierte Base die Viren-DNA vor dem Angriff bakterieller Verdauungsenzyme (Restriktionsendonucleasen) zu schützen [1].
Auch zu dieser Base finden Sie in der Wikipedia weitere wichtige Informationen.
5-Formylcytosin
Diese Cytosin-Modifikation wurde 2011 in embryonalen Stammzellen von Säugetieren entdeckt. Es handelt sich bei dieser Verbindung ebenfalls um einen epigenetischen Marker [2]. Gebildet wird 5-Formylcytosin durch Oxidation von 5-Hydroxymethylcytosin unter Katalyse von TET-Enzymen.
Die Forschung zu 5-Formylcytosin steckt noch in den Anfängen, daher kann man jetzt noch nicht allzu viel dazu sagen.
5-Carboxylcytosin
Zu dieser Modifikation gibt es noch nicht einmal einen Eintrag in der engl. Wikipedia, allerdings findet man viele Fachartikel (englischsprachig) im Internet.
Laut einem Artikel auf epigenie.com könnte 5-Carboxylcytosin das Ergebnis einer Demethylierung von 5-Methylcytosin sein. 5-Methylcytosin ist ein epigenetischer Marker, beeinflusst also die Transkriptionsrate der methylierten Gene. Durch die Umwandlung von 5-Methylcytosin in 5-Carboxylcytosin wird dieser epigenetische Marker wieder entfernt.
Überblick über die verschiedenen Modifikationen des Cytosins
In dem eben bereits erwähnten Wikipedia-Artikel "TET enzymes" findet sich eine sehr übersichtliche Graphik, die zeigt, in welchem Verhältnis die verschiedenen Cytosin-Modifikationen zueinander stehen:
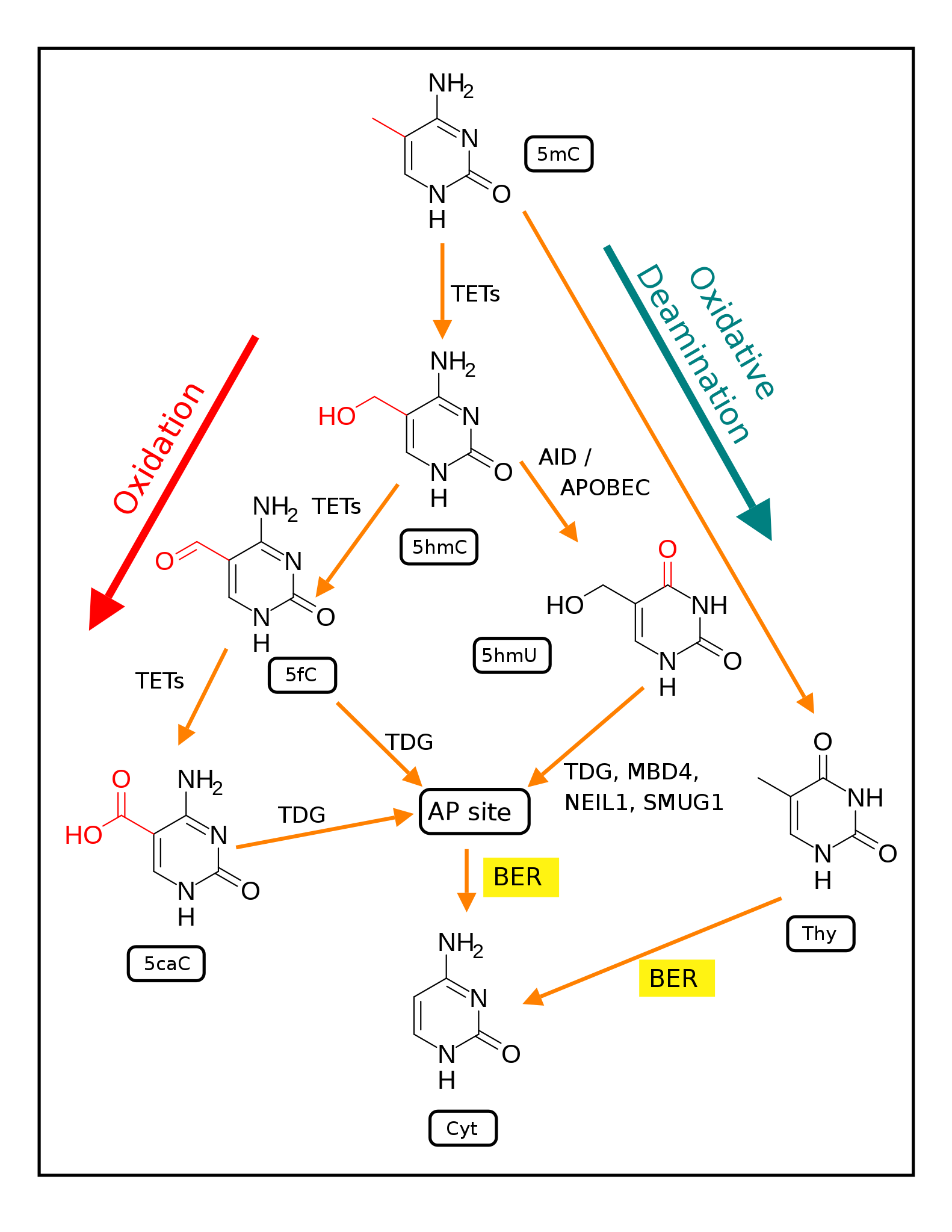
Zusammenhang zwischen den verschiedenen Cytosin-Modifikationen
Bernstein0275, User:Innerstream, CC BY-SA 4.0, via Wikimedia Commons
Der Reaktionsweg auf der linken Seite der Abbildung (Oxidation):
Oben im Bild sehen wir das 5-Methylcytosin-Molekül, das oft als 5mC abgekürzt wird. Durch Oxidation erhält man mit Hilfe der TET-Enzyme das 5-Hydroxymethylcytosin (5hmC), eine weitere TET-vermittelte Oxidation führt zu 5-Formylcytosin (5fC), und noch eine weitere, ebenfalls TET-katalysierte Oxidation liefert schließlich 5-Carboxylcytosin (5caC).
Der Reaktionsweg auf der rechten Seite der Abbildung (Desaminierung):
Das 5-Hydroxymethylcytosin wird durch bestimmte Enzyme desaminiert, das heißt, die Aminogruppe wird gegen ein Sauerstoff-Atom ausgetauscht, es entsteht dadurch 5-Hydroxymethyluracil, also eine ganz andere Base. Diese Uracil-Modifikation kann dann wieder in Cytosin (ganz unten im Bild) umgewandelt werden.
Ein weiterer Weg, ebenfalls rechts im Bild zu sehen, ist die direkte Desaminierung von 5-Methylcytosin zu Thymin, das dann ebenfalls leicht wieder in Cytosin umgewandelt werden kann.
Für alle, die lieber die Original-Bildbeschreibung lesen möchten, hier die engl. Fassung (Hervorhebungen von mir):
"Demethylation of 5-Methylcytosine (5mC) in DNA. As reviewed in 2018 5mC is oxidized by the ten-eleven translocation (TET) family of dioxygenases (TET1, TET2, TET3) to generate 5-hydroxymethylcytosine (5hmC). In successive steps TET enzymes further hydroxylate 5hmC to generate 5-formylcytosine (5fC) and 5-carboxylcytosine (5caC). Thymine-DNA glycosylase (TDG) recognizes the intermediate bases 5fC and 5caC and excises the glycosidic bond resulting in an apyrimidinic site (AP site).
In an alternative oxidative deamination pathway, 5hmC can be oxidatively deaminated by activity-induced cytidine deaminase/apolipoprotein B mRNA editing complex (AID/APOBEC) deaminases to form 5-hydroxymethyluracil (5hmU) or 5mC can be converted to thymine (Thy).
5hmU can be cleaved by TDG, single-strand-selective monofunctional uracil-DNA glycosylase 1 (SMUG1), Nei-Like DNA Glycosylase 1 (NEIL1), or methyl-CpG binding protein 4 (MBD4). AP sites and T:G mismatches are then repaired by base excision repair (BER) enzymes to yield cytosine (Cyt)."
Quellen:
- Wikipedia, Artikel "5-Methylcytosin" und "5-Hydroxymethylcytosin".
- engl. Wikipedia, Artikel "5-Formylcytosine"
- engl. Wikipedia, Artikel "TET enzymes"
- "5-carboxylcytosine (5caC)" auf epigenie.com.