Bakterien werden ständig von Viren bedroht, die ihre DNA oder RNA in die Bakterienzelle injizieren und den bakteriellen Proteinsynthese-Apparat so umprogrammieren, dass statt der Bakterienproteine neue Viren produziert werden.
Als Gegenmaßnahme haben die Bakterien im Laufe der Zeit Enzyme entwickelt, die virale DNA unschädlich machen - die Restriktionsenzyme. Eine spezielle Klasse dieser Restriktionsenzyme - die Restriktionsendonucleasen - zerschneidet die fremde DNA regelrecht. Die bakterieneigene DNA wird durch Methylierung der Basen gegen die eigenen Restriktionsenzyme geschützt (Maskierung); Viren-DNA trägt solche Methylgruppen meistens nicht und wird daher von den Restriktionsendonucleasen normalerweise als fremd erkannt. Einige Viren haben sich jedoch im Laufe der Zeit an diesen Maskierungsmechanismus angepasst und tragen ebenfalls Methylgruppen auf der DNA, so dass sie von den bakteriellen Restriktionsendonucleasen nicht mehr angegriffen werden können.
In der Gentechnik kennt man inzwischen viele verschiedene Restriktionsendonucleasen. Jede Restriktionsendonuclease schneidet die DNA an einer spezifischen Basensequenz, der Restriktionsschnittstelle, die meistens aus vier bis acht Basenpaaren besteht. Die Restriktionsendonuclease in der folgenden Abbildung hat zum Beispiel die Basensequenz TAAG als Restriktionsschnittstelle.
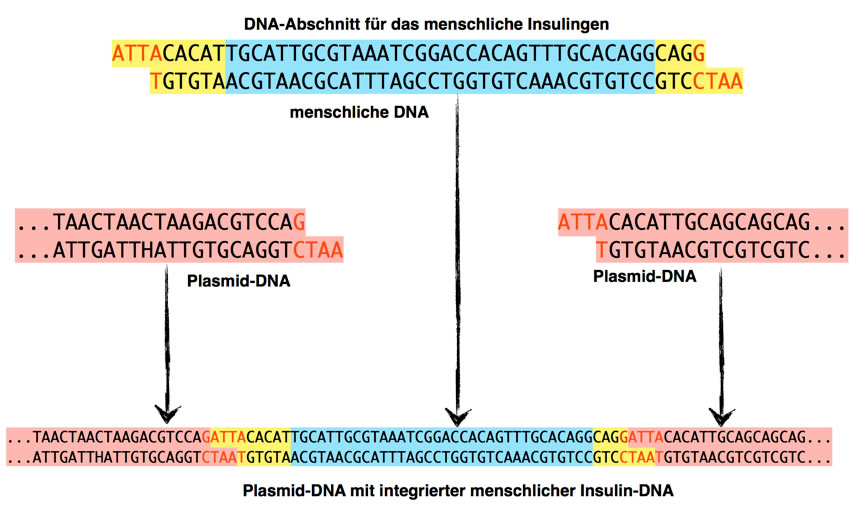
Mit Restriktionsendonucleasen kann man DNA-Stränge passgenau aufschneiden
Das Bild oben zeigt ein menschliches Insulingen, das mit einer Restriktionsendonuclease aus der menschlichen DNA herausgeschnitten wurde. Man achte auf die "sticky ends", die "klebrigen Enden" links und rechts des Insulin-Gens. Diese sticky ends sind eine Folge des Herausschneidens. Die meisten Restriktionsendonuclease schneiden die DNA nämlich nicht einfach quer durch, sondern versetzt, so dass ein Überhang von zwei bis vier Nucleotiden entsteht. In unserem Beispiel besteht dieser Überhang aus je drei Nucleotiden.
Ein bakterielles Plasmid wird mit der gleichen Restriktionsendonuclease aufgeschnitten, so dass die gleichen sticky ends entstehen. Wie man leicht sieht, sind die überhängenden Enden des aufgeschnittenen Plasmides komplementär zu den überhängenden Enden des Insulin-Gens, so dass dieses leicht in das geöffnete Plasmid eingebaut werden kann.
Drei wichtige Restriktionsendonucleasen
Hae III
Diese Restriktionsendonuclease von Haemophilus aegyptius erkennt eine Sequenz aus vier Basen, nämlich 5'-GGCC-3' und schneidet die DNA senkrecht durch. So geschnittene DNA kann man nicht so leicht in Plasmide oder andere Vektoren einbauen, daher ist die Bedeutung für die Gentechnik eher gering.
Taq I
Diese Restriktionsendonuclease erkennt ebenfalls eine Sequenz aus vier Basen, nämlich 5'-TCGA-3', schneidet die DNA aber versetzt durch mit einem Überhang von zwei Basen.
Eco RI
Diese Restriktionsendonuclease von Escherichia coli erkennt eine Sequenz aus sechs Basen, nämlich 5'-GAATTC-3' und schneidet die DNA versetzt mit einem Überhang von vier Basen durch:
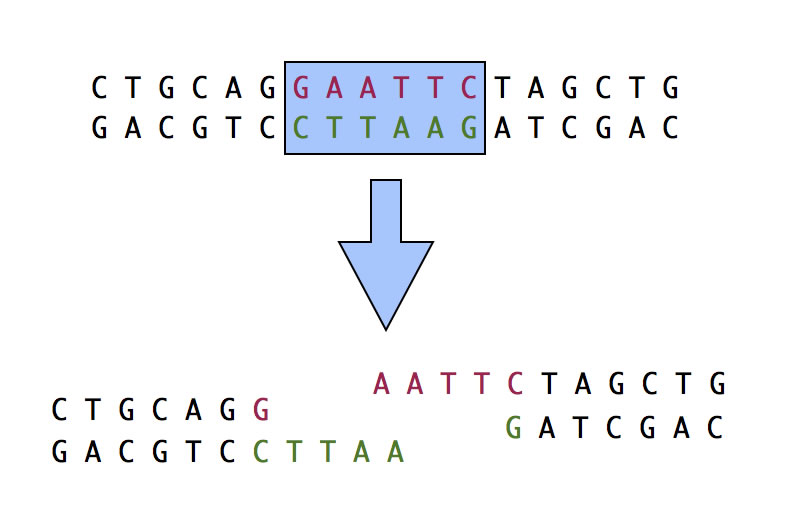
EcoRI schneidet die DNA mit einem Überhang von vier Nucleotiden durch
Dieses Restriktionsenzym ist sehr gut für gentechnische Zwecke geeignet und wird daher auch sehr oft eingesetzt.