Die DNA-Polymerase III ist das Schlüsselenzym bei der Replikation der DNA, leistet also die Hauptarbeit. Diese besteht darin, ein neues Nucleotid an einen wachsenden DNA-Einzelstrang anzuhängen und anschließend eventuell eine Fehlerkorrektur durchzuführen, also ein falsch eingebautes Nucleotid sofort wieder zu entfernen, bevor das nächste Nucleotid eingebaut wird.
Entdeckt wurden die DNA-Polymerasen durch Arthur Kornberg 1953 (DNA-Polymerase I) sowie durch seine Söhne Roger und Thomas Kornberg 1970 (DNA-Polymerasen II und III).
Arbeitsweise
Die folgende Zeichnung verdeutlicht die Arbeitsweise der DNA-Polymerase III:
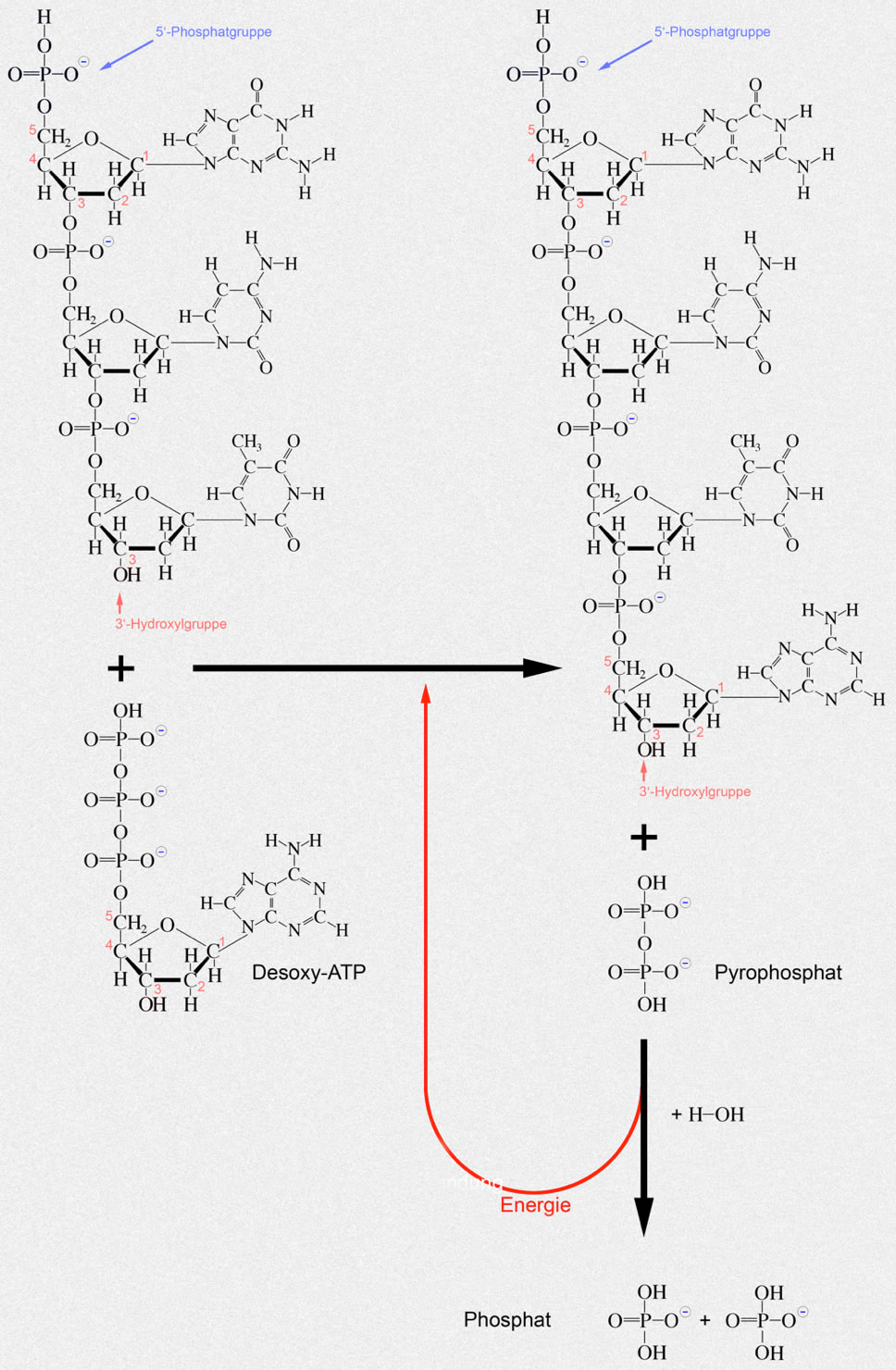
Elementarreaktion der DNA-Polymerase III
Autor: Ulrich Helmich 2018, Lizenz: Public domain
Die C3-OH-Gruppe am 3'-Ende des DNA-Einzelstrangs greift das innere Phosphor-Atom des neuen Nucleotids (hier: Desoxy-ATP) nucleophil an; das Phosphor-Atom des neuen Nucleotids verbindet sich kovalent mit dem O-Atom der eben erwähnten OH-Gruppe. Zwei Phosphatreste werden als Pyrophosphat abgespalten. Beim Zerfall des Pyrophosphats in zwei Phosphatreste wird ein hoher Energiebetrag freigesetzt, welcher der gesamten Reaktion wieder zugute kommt.
Wichtig sind hier vor allem zwei Punkte
- Die DNA-Polymerase III kann die neuen Nucleotide immer nur an das 3'-Ende des DNA-Einzelstrangs anhängen.
- Die DNA-Polymerase kann nicht bei Null starten, sie braucht immer einen sogenannten Primer, an dessen 3'-OH-Ende sie das neue Nucleotid anhängen kann.
- Die DNA-Polymerase III verfügt über eine Nuclease-Aktivität. Sie kann also falsch eingebaute Nucleotide sofort wieder entfernen, bevor sie das nächste Nucleotid einbaut.
Dieser Primer kann eine einsträngige DNA oder auch eine RNA sein. Bei der DNA-Replikation der Pro- und Eukaryoten ist dieser Primer stets ein kurzes RNA-Stück, vielleicht so um die 10 Nucleotide lang.
Struktur
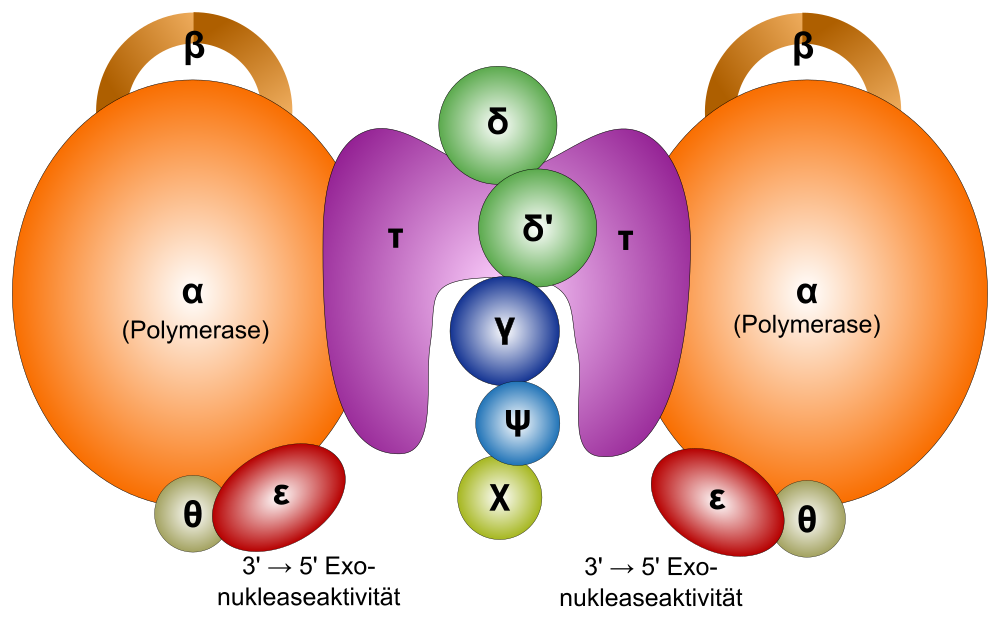
Der Multienzymkomplex DNA-Polymerase III
Quelle: Wikipedia, Artikel "DNA-Polymerase III" , Stand: 15.12.2021, Autor: Yikrazuul, Lizenz: public domain.
Interessanterweise findet man bei einer Recherche zur Struktur der DNA-Polymerase III sehr unterschiedliche Darstellungen in der Fachliteratur und im Internet. Oben sehen wir ein Bild aus dem Artikel "DNA-Polymerase III" in der deutschen Wikipedia. In der englischsprachigen Wikipedia findet sich eine völlig andere Darstellung, wie der folgende Screenshot zeigt:
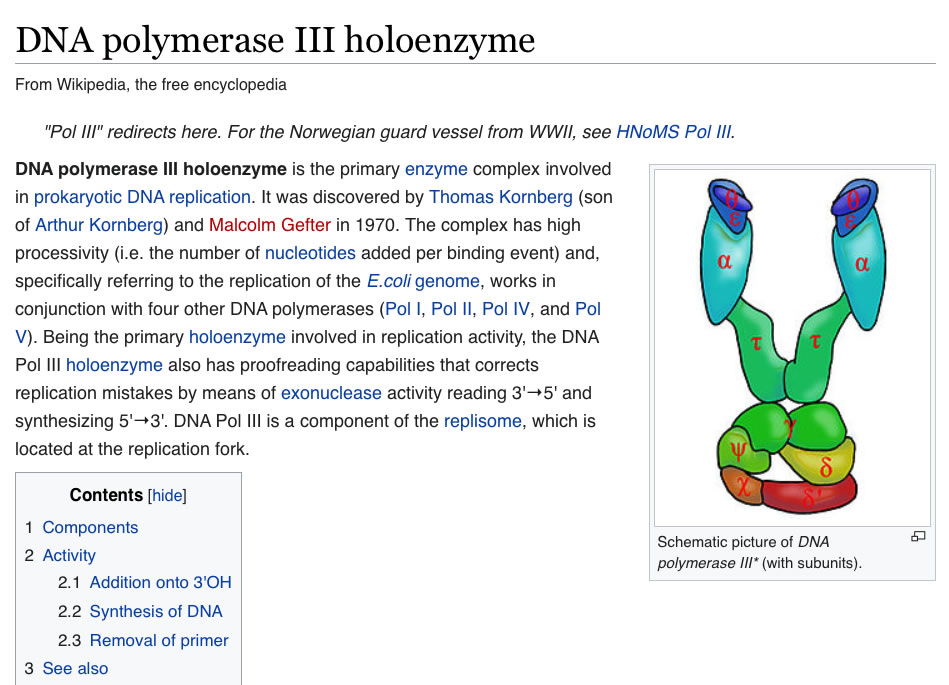
Screenshot des Artikels der engl. Wikipedia, erstellt am 17.12.2021.
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Und in meinem alten Darnell, Lodisch, "Molekulare Zellbiologie" aus dem letzten Jahrtausend habe ich folgendes Bild gefunden:

Darstellung der DNA-Polymerase III im Darnell, Lodish, Molekulare Zellbiologie von 1996
Am interessantesten ist das Bild der Polymerase in dem neuen Lehninger [3]:

Darstellung der DNA-Polymerase III im neuen Lehninger, Biochemistry von 2021
Nach dem Lehninger besteht die DNA-Polymerase III aus vier großen Abschnitten. Drei davon werden als Pol III core bezeichnet, eine als Clamp loader. Ich will jetzt mal versuchen, diese Darstellung zu vereinfachen und dann mit meinen eigenen Worten zu erklären. Hier das Ergebnis meiner bescheidenen Versuche:
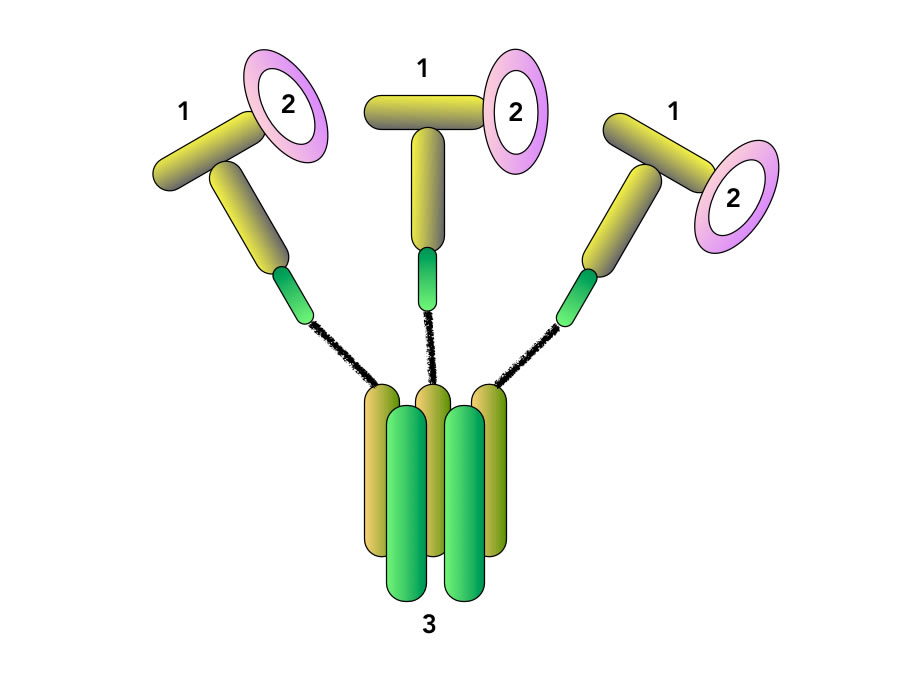
Die bakterielle DNA-Polymerase III in Anlehnung an die Zeichnung im neuen Lehninger von 2021
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Welche Aufgaben haben nun die einzelnen Untereinheiten der DNA-Polymerase III ? Ich habe mich hier auf das Wesentliche beschränkt und unterscheide nur drei Typen von Untereinheiten. Jede Untereinheit besteht ihrerseits wieder aus zwei, drei oder mehr Untereinheiten.
Untereinheit 1: Pole III core
Diese Untereinheit ist für die Elementarreaktion der Replikation verantwortlich; das Anhängen eines neuen Nucleotids an den wachsenden DNA-Einzelstrang.
Untereinheit 2: β sliding clamp
Dieser aus zwei Untereinheiten bestehende Ring umschließt den jeweiligen DNA-Einzelstrang und leitet ihn zu der Untereinheit 1.
Untereinheit 3: Clamp loader
Dieser aus fünf Untereinheiten bestehende Komplex klammert (clamp = Klammer) im Grunde alles zusammen.
Arbeitsweise der Polymerase
Im Gegensatz zu allen anderen Darstellungen in der Literatur und im Internet hat die Lehninger-Polymerase drei aktive Untereinheiten, die DNA-Stränge verlängern können. Im weiteren Text wird dann erläutert, dass eine dieser drei Pole III cores für die DNA-Synthese am Leitstrang verantwortlich ist, während die beiden anderen Poly III cores zwei Okazaki-Fragmente gleichzeitig synthetisieren. Während die eine Untereinheit mit ihrem Fragment langsam fertig wird, beginnt die andere Untereinheit schon das neue Okazaki-Fragment nahe der Replikationsgabel zu synthetisieren.
Ich habe mal versucht, das Ganze zeichnerisch darzustellen. Die folgende Graphik ist ein Digital-Analog-Hybrid. Am PC wurden die Proteine in der richtigen Orientierung gezeichnet, die Graphik wurde dann ausgedruckt und mit Hand habe ich dann die DNA-Stränge eingezeichnet und dabei auch die Schleifenbildung des lagging strands berücksichtigt. Anschließend habe ich das Blatt wieder eingescannt und dann mit Photoshop leicht korrigiert und danach beschriftet. Hier das Ergebnis meiner Bemühungen:
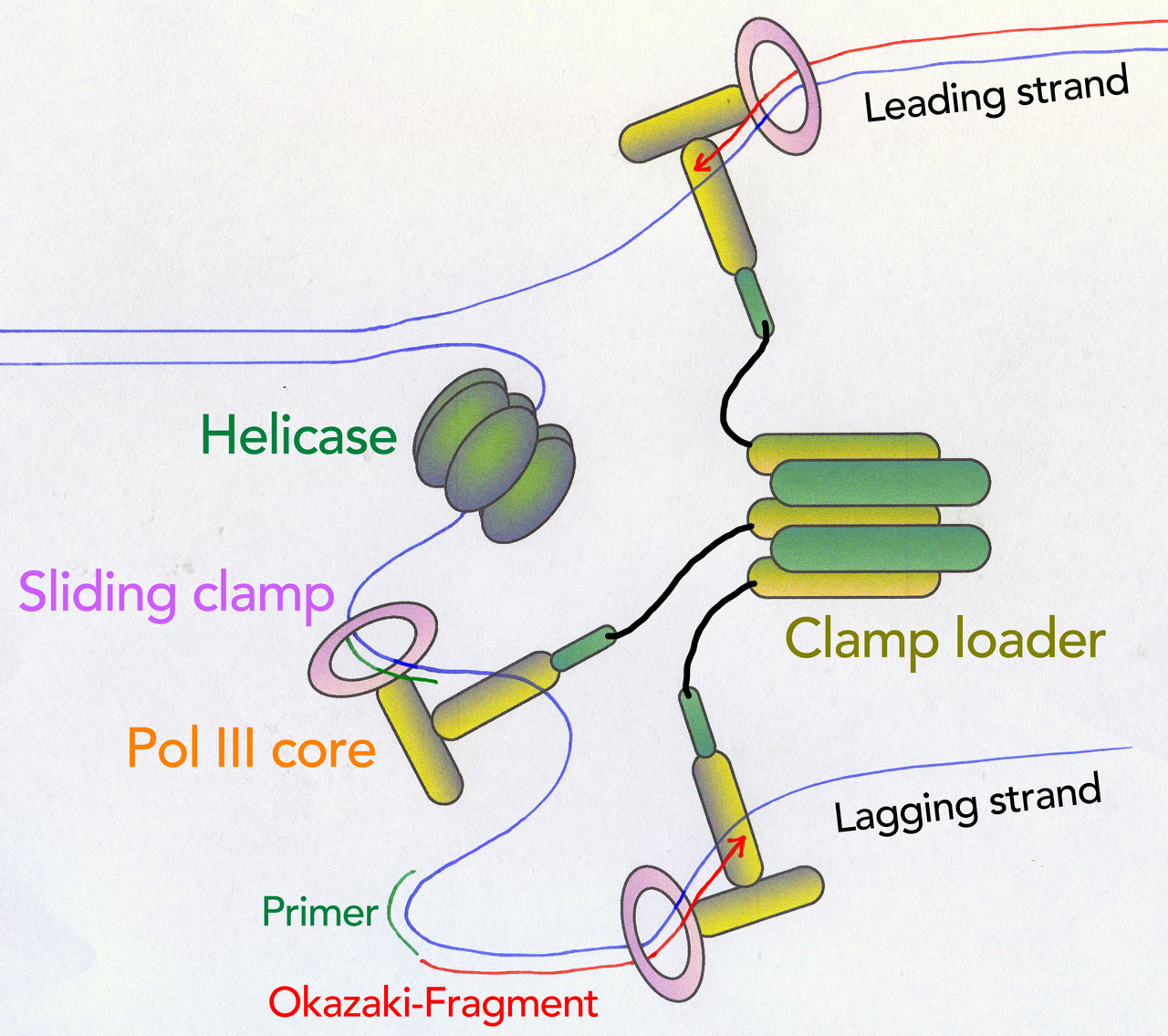
Die DNA-Polymerase III in Aktion
Autor: Ulrich Helmich 2021, nach Figure 25-11 im aktuellen Lehninger, Lizenz: Public domain
Die ringförmige Sliding clamp (Gleitklammer) umschließt den DNA-Einzelstrang und führt ihn zum Pol III core (also zur Haupteinheit der Polymerase III). Der mittlere Pol III core umschließt gerade den soeben fertiggestellten RNA-Primer (die Primase ist schon wieder wegdiffundiert, daher sieht man sie hier nicht), und der untere Pol III core ist gerade damit beschäftigt, ein Okazaki-Fragment herzustellen. Der obere Pol III core arbeitet an dem Leading strand, hier läuft die Replikation ab wie "geschmiert", also schulbuchartig ohne Probleme. Man achte auf die Helicase, diese befindet sich grundsätzlich am Lagging strand. Das ganze Gebildet bewegt sich nach links weiter, vielleicht hätte ich da noch einen dicken Pfeil einzeichnen sollen, um das noch deutlich er zu machen.
Wirklich drei Core-Einheiten?
Der Lehninger ist ja ein tolles Buch, er hat mich seit dem Studium in den 80er Jahren immer treu begleitet. Aber hat er wirklich recht, wenn er in seiner neuesten Auflage von 2021 behauptet, dass die DNA-Polymerase III nicht zwei, sondern drei Core-Einheiten besitzt? Er schreibt sogar auf Seite 925:
"DNA polymerase III uses one set of its core subunits ... to synthesize the leading strand continuously, while the other two sets of core subunits cycle from one Okazaki fragment to the next on the looped lagging strand."
Prinzipiell käme die Polymerase auch mit zwei Core-Units aus, doch:
"However, a third set of core subunits increases the efficiency of lagging strand synthesis as well as the processivity of the overall replisome."
Das hört sich doch alles sehr überzeugend an. Wenn wir dagegen eine Bildersuche im Internet machen mit dem Stichwort "DNA-Polymerase III", dann sehen wir folgendes:
Ergebnis einer Google-Bildersuche am 18.12.2021
Auf keinem Bild findet man eine Polymerase III mit drei Core-Einheiten. Auch die Fachliteratur stellt die Polymerase normalerweise mit nur zwei Core-Einheiten dar, zumindest die Bücher, die ich selbst besitze (siehe Quellen).
Quellen:
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Rolf Knippers: Molekulare Genetik, 9. Auflage, Thieme-Verlag Stuttgart 2006.
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.
- Kadereit , Körner, Nick, Sonnewald: Strasburger - Lehrbuch der Pflanzenwissenschaften, 38. Auflage, Springer Berlin Heidelberg 2021.
- Urry, Cain, Wassermann, Minorsky, Reece. Campbell Biologie, Hallbergmoos 2019, 11.Auflage.
- Plattner, Hentschel. Zellbiologie, 5. Auflage. Stuttgart 2017.