Lernziele
Wenn Sie diese Seite durchgearbeitet haben, sollten Sie wissen
- dass DNA die Abkürzung für "Deoxyribonucleic acid" ist,
- dass die DNA aus zwei Einzelsträngen besteht, die sich umeinander winden, und dass man diese Anordnung als "Doppelhelix" bezeichnet.
- wie jeder Einzelstrang chemisch aufgebaut ist, nämlich aus den drei Bausteinen Desoxyribose, Phosphorsäure und einer von vier DNA-Basen,
- wie die Strukturformeln von Phosphorsäure und Desoxyribose (ungefähr) aussehen,
- wie die vier DNA-Basen ungefähr aufgebaut sind
- was man unter dem 3'-Ende und dem 5'-Ende der Einzelstränge versteht,
- wie das Prinzip der Basenpaarung funktioniert,
- dass zwischen A und T zwei H-Brücken existieren, zwischen G und C dagegen drei,
- welche Bedeutung die Basenpaarung für den Aufbau der DNA hat.
Kurzfassung
Wenn Ihnen diese Seite hier zu lang oder zu kompliziert ist, können Sie sich auch die Kurzfassung der Seite anschauen.
Chemische Struktur der DNA
Bereits Friedrich Miescher (1844 - 1895) hat 1869 bis 1871 in seinem Labor die chemischen Verbindungen entdeckt, die man heute als Nucleinsäuren bezeichnet. Miescher nannte die Substanz, die er im Eiter aus Verbandsmaterial gefunden hatte, Nuklein. Auch im Sperma von Lachsen fand er diese Verbindung. Miescher fiel auch auf, dass das Nuklein sehr viel Phosphat enthielt. Aber insgesamt war das Nuklein für Miescher eine eher langweilige Substanz, und kurze Zeit nach der Entdeckung wandte er sich den Proteinen zu, sie sehr vielversprechender waren, da sie aus 20 verschiedenen Aminosäuren aufgebaut waren. Das war doch deutlich abwechslungsreicher als die Nucleinsäuren.
Der Begriff "Nucleinsäure" wurde übrigens erst 1899 von Richard Altmann eingeführt, obwohl bereits Miescher erkannt hat, dass das sein Nuklein saure Eigenschaften hatte.
Schauen wir uns nun den chemischen Aufbau der DNA etwas näher an. Die DNA liegt in Form einer Doppelhelix vor, die aus zwei gegenläufigen Strängen besteht. Jeder DNA-Einzelstrang setzt sich aus drei verschiedenen Komponenten zusammen:
- Phosphorsäure
- Desoxyribose, eine Pentose (d.h. ein Zucker mit 5 C-Atomen)
- Einer von vier möglichen DNA-Basen
Im Folgenden wollen wir diese drei Bausteine näher kennenlernen.
Baustein 1: Phosphorsäure
Schon die chemische Analysen von Mitscherlich Ende des 19. Jahrhunderts ergaben, dass die DNA sehr viel Phosphorsäure-Reste enthält. Schauen wir uns mal ein Molekül der Phosphorsäure H3PO4an.
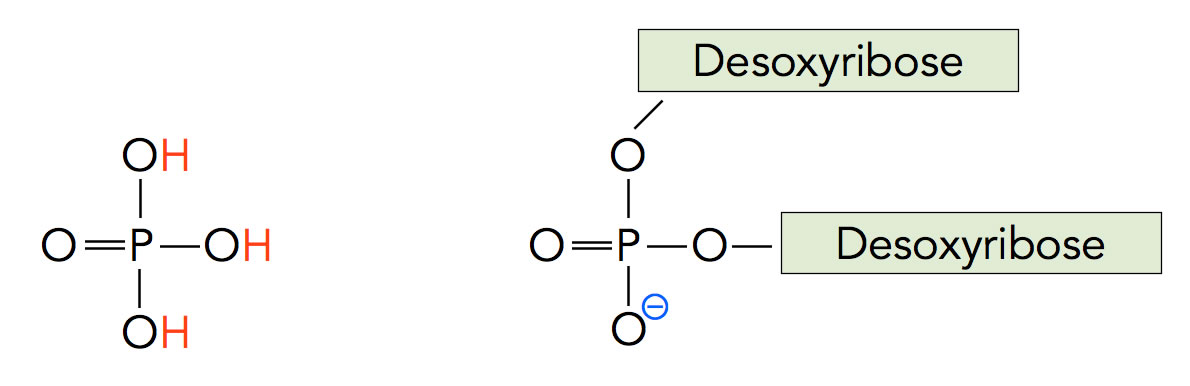
Ein Phosphorsäure-Molekül (links) und eine Phosphatgruppe, die mit zwei Desoxyribose-Molekülen verbunden ist.
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Die Phosphorsäure ist eine dreiprotonige Säure. Wird nur ein Proton abgegeben, entsteht das Dihydrogenphosphat-Ion H2PO4-. Nach Abgabe des zweiten Protons bildet sich das Hydrogenphosphat-Ion HPO42-. Wenn dann das dritte Proton abgegeben wurde, liegt die Phosphorsäure als Phosphat-Ion PO43- vor. So viel zum Säure-Verhalten der Phosphorsäure.
Die drei OH-Gruppen können aber noch mehr als nur ein Proton abzugeben. Sie können sich nämlich mit anderen OH-Gruppen verbinden, zum Beispiel mit OH-Gruppen aus Zucker-Molekülen. Die Desoxyribose ist ein solcher Zucker. In der DNA verbindet sich jedes Phosphorsäure-Molekül sogar mit zwei Desoxyribose-Molekülen.
Baustein 2: Desoxyribose
Man fand schnell heraus, dass die DNA nicht nur viele Phosphorsäure-Bausteine enthält, sondern auch exakt so viel Desoxyribose-Bausteine. Desoxyribose ist ein Monosaccharid mit fünf C-Atomen. Hier eine Zeichnung dieses Zuckers.
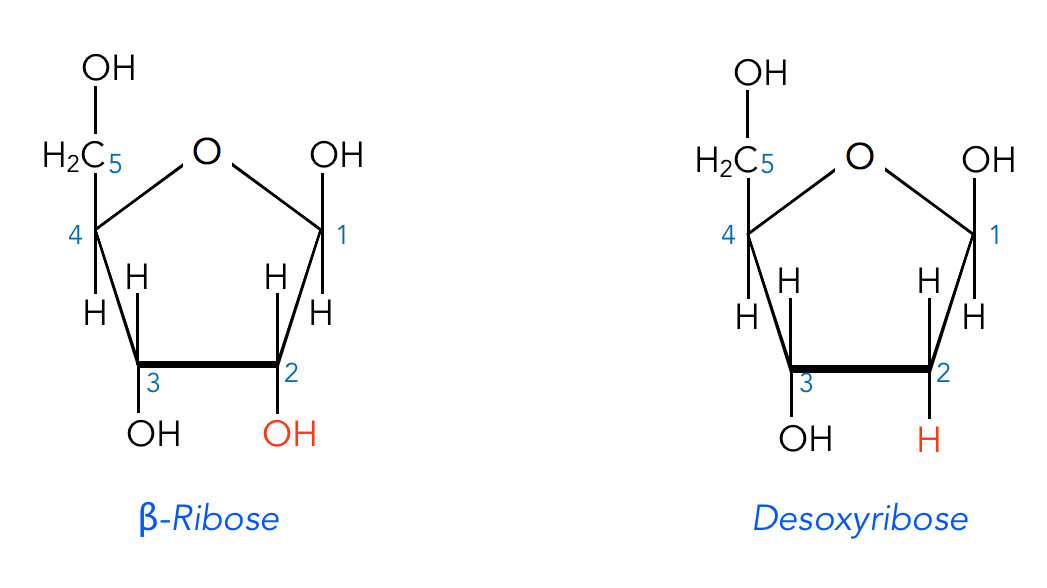
Ein β-Ribose- und ein Desoxyribose-Molekül
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Streng genommen ist die Desoxyribose gar kein Kohlenhydrat, sondern ein chemisch verändertes Kohlenhydrat. Die Desoxyribose ist nämlich die reduzierte Form des Monosaccharids Ribose. Die OH-Gruppe am C2-Atom wurde durch ein einfaches H-Atom ersetzt. Es bleiben nur noch drei OH-Gruppen an den Kohlenstoff-Atomen 1, 3 und 5, und mit diesen drei OH-Gruppen kann sich die Desoxyribose mit anderen Molekülen verbinden:
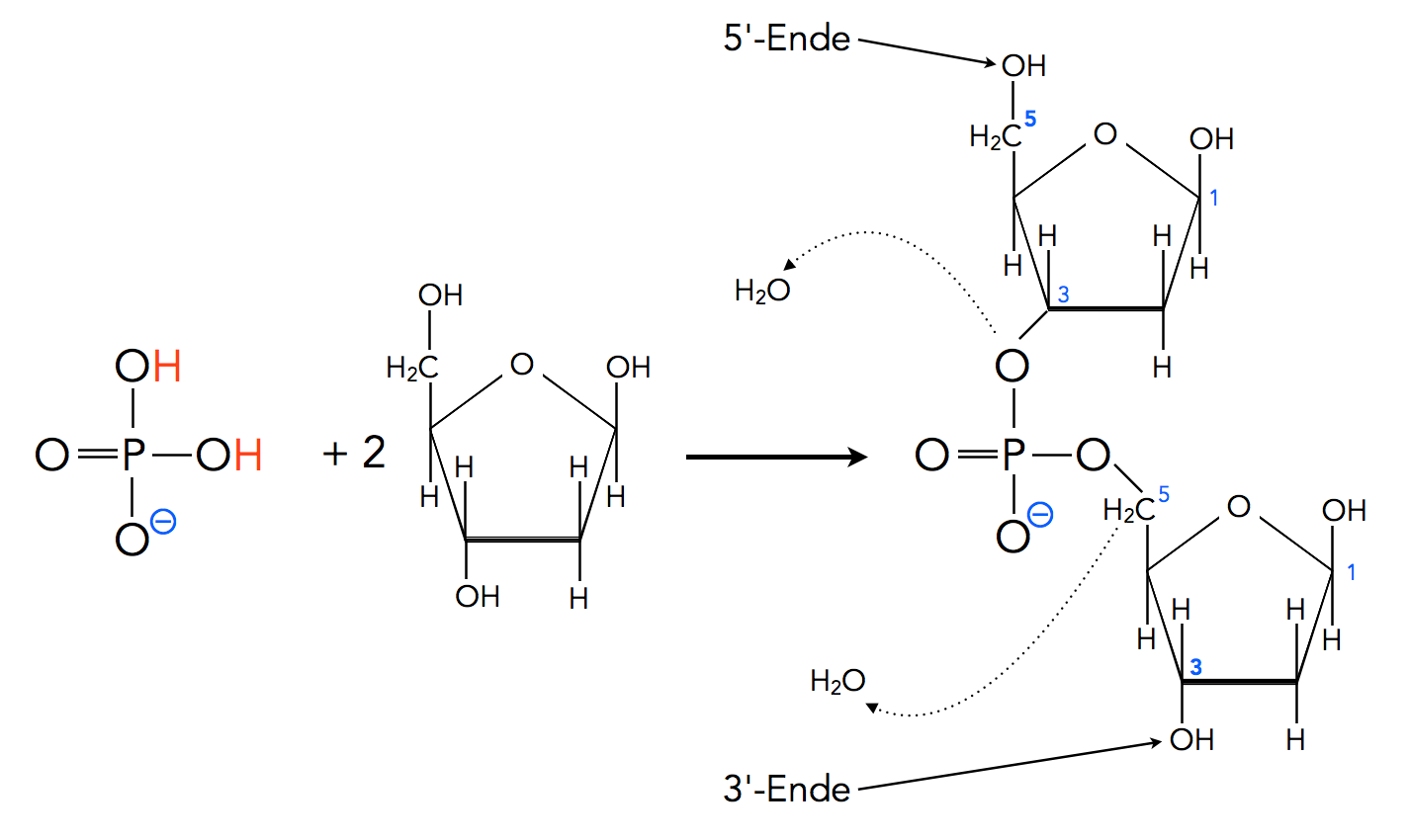
Ein Phosphorsäure-Rest (Dihydrogenphosphat-Ion) reagiert mit zwei Desoxyribose-Molekülen unter Wasser-Abgabe
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Auf diesem Bild sehen wir, wie sich ein Dihydrogenphosphat-Ion (manche Leute sagen auch einfach "ein Phosphat-Rest") mit zwei Desoxyribose-Molekülen verbindet. Ein O-Atom des Phosphat-Restes verbindet sich mit dem C3-Atom des einen Desoxyribose-Moleküls, und ein anderes O-Atom des Phosphat-Restes verbindet sich mit dem C5-Atom des zweiten Desoxyribose-Moleküls.
Das im Bild oben stehende Desoxyribose-Molekül hat noch eine intakte OH-Gruppe am C5-Atom, daher wird dieses Ende des Gesamtmoleküls als 5'-Ende bezeichnet (gelesen: 5 - Strich - Ende). Entsprechend hat das "untere" Desoxyribose-Molekül eine intakte OH-Gruppe am C3-Atom, daher bezeichnet man dieses Ende als 3'-Ende.
Dieses große Molekül aus einem Phosphat-Rest und zwei Ribose-Resten ist natürlich noch kein DNA-Einzelstrang. Aber die beiden Ribose-Reste haben ja noch zwei freie, unbesetzte OH-Gruppen an den C-Atomen 1 und 5 (oberer Ribose-Rest) bzw. 1 und 3 (unterer Ribose-Rest). Mit diesen OH-Gruppen können sich die Ribose-Reste nun mit anderen Molekülen verbinden, wobei ebenfalls Wasser abgespalten wird.
Wenn sich die Phosphorsäure mit einer Desoxyribose verbindet, ist das chemisch gesehen eine Veresterung. Mehr zu diesem Vorgang und der Stoffgruppe der Ester erfahren Sie auf dieser Seite im Chemie-Lexikon.
Schauen wir uns dazu das nächste Bild an:
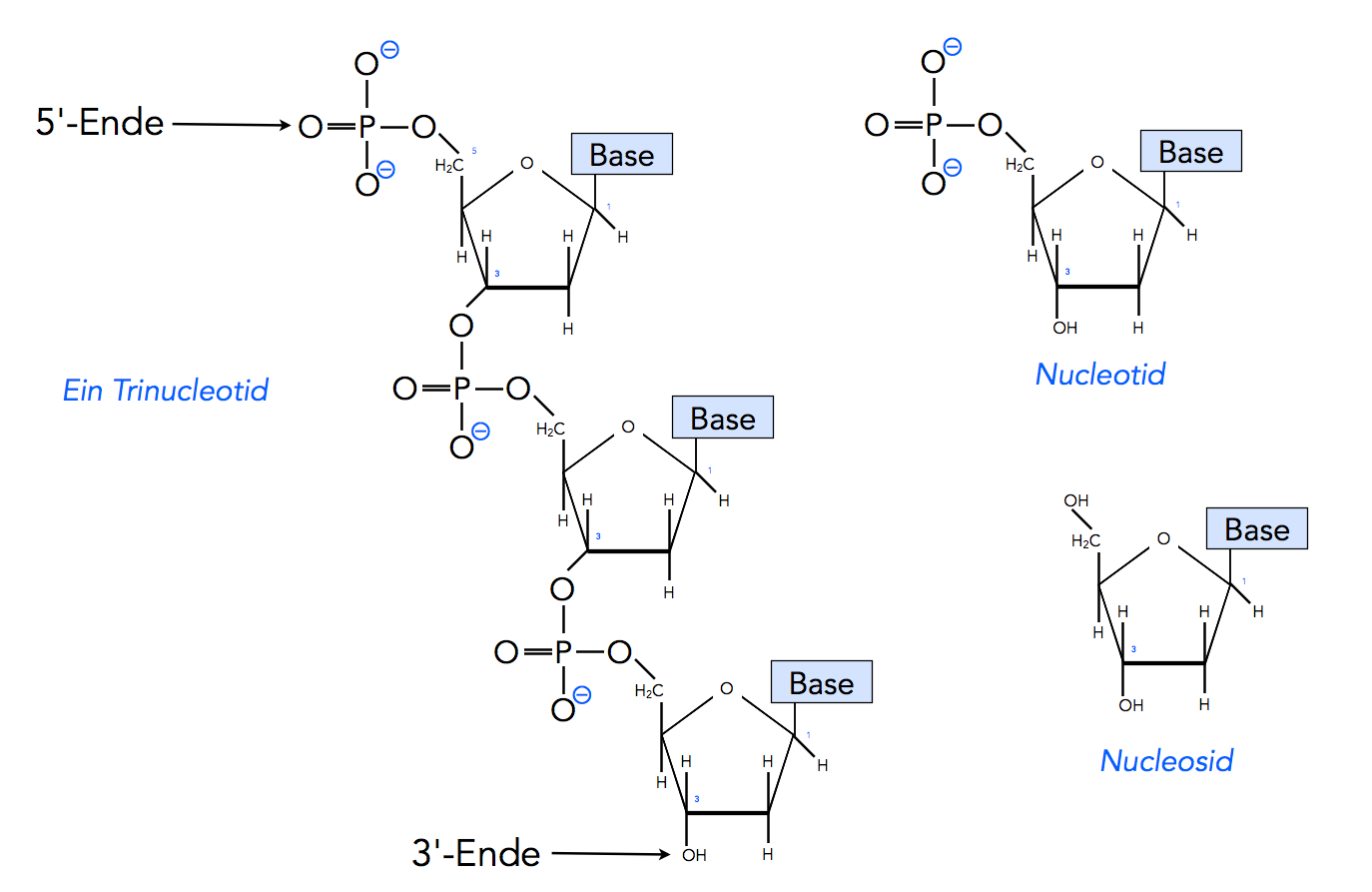
Ein Trinucleotid sowie ein Nucleotid und ein Nucleosid
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Hier sehen wir im linken Bereich schon ein etwas längeres Molekül, das aus drei Phosphat-Resten und drei Desoxyribose-Resten besteht. Auch dieser Strang hat ein 5'-Ende (oben im Bild) und ein 3'-Ende (unten im Bild), kann also in beide Richtungen noch wachsen, indem die Phosphat-Reste weitere Desoxyribose-Reste anlagern und die Desoxyribose-Reste weitere Phosphat-Rest.
Ein paar Fachbegriffe, die man kennen sollte
Nucleotid
Unter einem Nucleotid versteht man ein Molekül, das aus einem Desoxyribose-Rest besteht, der am C-Atom 5 mit einem Phosphat-Rest und am C-Atom 3 mit einer Base verbunden ist. Diese Basen behandeln wir im nächsten Abschnitt auf dieser Seite.
Nucleosid
Ein Nucleosid ist ein Nucleotid ohne den Phosphatrest.
Trinucleotid
Ein Trinucleotid sehen wir in der Abbildung. Es besteht aus drei miteinander verknüpften Nucleotiden.
DNA-Einzelstrang
Ein DNA-Einzelstrang ist ein Polynucleotid, besteht also aus vielen, teils mehreren Tausend miteinander verknüpften Nucleotiden.
Baustein 3: Die Basen
Kommen wir nun zu der entscheidenden Frage, was die beiden Einzelstränge der DNA eigentlich zusammenhält. Watson und Crick, die beiden berühmten Entdecker der DNA-Struktur, hatten damit am Anfang ziemliche Probleme. Das Rückgrat beider Einzelstränge ist stark negativ geladen, jeder Phosphat-Rest trägt ja eine negative Ladung. An sich müssten sich die beiden DNA-Stränge daher gegenseitig abstoßen.
Für Chemie-Experten
Das ist natürlich nur dann der Fall, wenn in dem umgebenden Medium ein Protonenmangel herrscht. Würde man eine Säure zu einer DNA-Aufbereitung dazugegeben, würden sich Protonen von den Säure-Molekülen abspalten und an die negativ geladenen Phosphat-Reste setzen. Daher ist die Struktur der DNA abhängig vom pH-Wert. Und weil die DNA-Struktur vom pH-Wert abhängig ist, darf sich in den Zellen der Lebewesen dieser pH-Wert auch nicht merklich verändern. Dafür sorgen dann verschiedene Puffersysteme in den Zellen und in der Körperflüssigkeit.
Wie sehen nun die vier DNA-Basen aus?
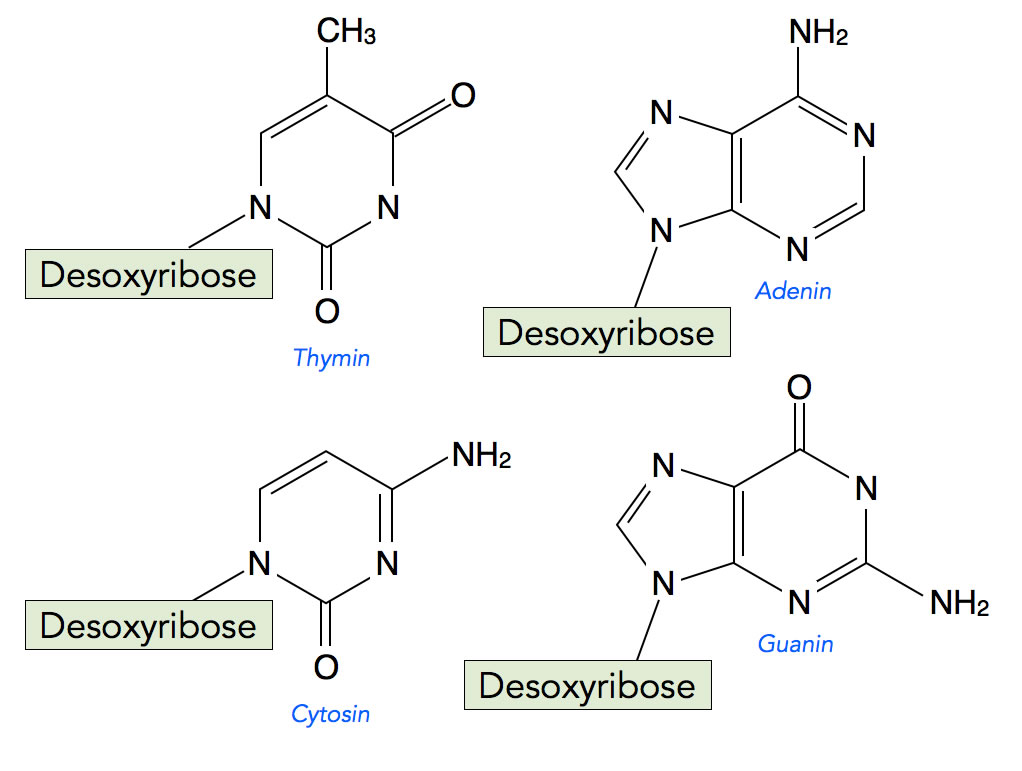
Die vier Basen der DNA, im Uhrzeigersinn: Thymin, Adenin, Guanin und Cytosin.
Autor: Ulrich Helmich 2021, Lizenz: Public domain.
Auf dem Bild sehen wir die vier DNA-Basen. Die beiden großen Basen Adenin und Guanin leiten sich von der Verbindung Purin ab. Purin besteht aus einem sechsgliedrigen Ring aus vier C-Atomen und zwei N-Atomen, der mit einem fünfgliedrigen Ring verbunden ist, der ebenfalls zwei N-Atome enthält.
Die beiden kleinen Basen Thymin und Cytosin leiten sich von der Verbindung Pyrimidin ab. Pyrimidin bestehen aus einem Ring aus vier C-Atomen und zwei N-Atomen.
An diesen Purin- und Pyrimidin-Ringen sitzen jetzt noch weitere Gruppen, am Adenin zum Beispiel eine Aminogruppe (-NH2), am Guanin eine Aminogruppe und ein doppelt gebundenes O-Atom, am Thymin zwei doppelt gebundene O-Atome sowie eine Methylgruppe, und am Cytosin ein doppelt gebundenes O-Atom sowie eine Aminogruppe.
Alle vier Basen der DNA sind mit dem C1-Atom der Desoxyribose verbunden, wie schon in Abbildung 4 dargestellt. Achten Sie bitte auf die Tatsache, dass Adenin nahezu die gleiche Größe hat wie Guanin, und Thymin die gleiche Größe wie Cytosin. Das spielt eine wichtige Rolle bei der Struktur der DNA-Doppelhelix, wie wir gleich noch sehen werden.
Auf dieser Lexikonseite finden Sie weitere Informationen zu den einzelnen DNA-Basen. Es werden auch zusätzliche Basen wie Uracil (RNA) oder Thymin-Dimere vorgestellt.
Basenpaarung
Was die beiden Einzelstränge zusammenhält, haben wir aber immer noch nicht geklärt, und damit treten wir in die Fußstapfen von James Watson (1928 geb.) und Francis Crick (1916-2004). Die wussten nämlich zuerst auch nicht, was die DNA eigentlich zusammenhält, weil beide recht wenig Ahnung von Chemie hatten. Die beiden Forscher haben die Basenpaarung erst ganz am Ende erkannt, nachdem sie zunächst ein föllig valsches Modell der DNA gebaut und sich damit heftig vor konkurrierenden Wissenschaftlern wie Rosalind Franklin oder Maurice Wilkins blamiert hatten, die zu der Zeit ebenfalls an der Struktur der DNA arbeiteten.
Die A-T-Paarung
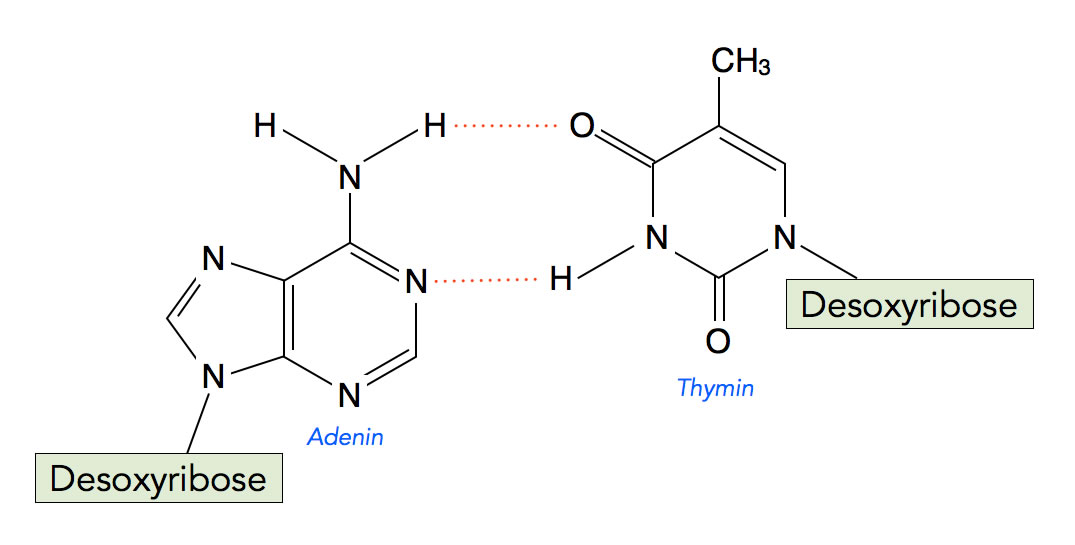
Das Basenpaar Adenin-Thymin
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Adenin und Thymin können sich mit Hilfe von zwei H-Brücken paaren. Um das zu verdeutlichen, wurden in der Abbildung einige H-Atome eingezeichnet, die man normalerweise nicht mit zeichnet, zum Beispiel an der NH2-Gruppe des Adenins. Die H-Brücken sind ungefähr gleich lang, knapp 0,3 nm.
Die G-C-Paarung
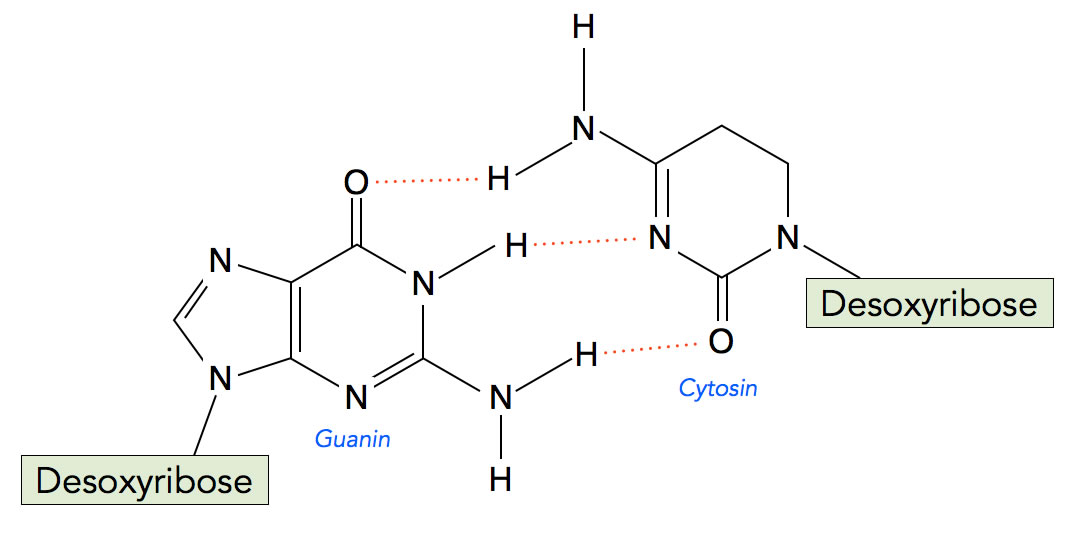
Das Basenpaar Guanin-Cytosin
Autor: Ulrich Helmich 2021, Lizenz: Public domain
Guanin und Cytosin können sich mit Hilfe von drei H-Brücken paaren. Auch hier sind die H-Brücken ungefähr gleich lang, knapp 0,3 nm.
Die Bindung zwischen Guanin und Cytosin ist deutlich stärker als die zwischen Adenin und Thymin, schließlich ist hier eine H-Brücke mehr am Werk. Außerdem werden die beiden komplementären DNA-Basen nicht ausschließlich durch H-Brücken zusammengehalten, auch van-der-Waals-Kräfte spielen eine Rolle. Auf diese wird aber in der Literatur meistens nicht eingegangen, weil sie viel schwächer sind als die Wasserstoffbrücken-Bindungen.
Bedeutung der Basenpaarung
Die vier Basen sind die vier Buchstaben des genetischen Codes. Die Reihenfolge der Basen auf dem einen DNA-Strang, dem so genannten codogenen Strang, stellt die eigentliche Erbinformation dar. Der andere Strang, der nicht-codogene Strang, ist quasi eine komplementäre Kopie des codogenen Strangs.
Dieses Prinzip der Komplementarität macht erst dann so richtig Sinn, wenn die DNA-Doppelhelix verdoppelt werden soll. Dann teilt sich nämlich die Doppelhelix an einer Stelle (dem Replikationsursprung) in zwei einzelne Stränge, die Basenpaare lösen sich voneinander, und anschließend sorgen viele spezielle Enzyme dafür, dass die jetzt ungepaarten Basen der beiden Einzelstränge wieder ergänzt werden. An jedes ungepaarte Adenin setzt sich also ein Thymin-Nucleotid, an jedes ungepaarte Guanin dockt ein Cytosin-Nucleotid an. Die so angedockten Nucleotide werden dann zu einem neuen Einzelstrang verknüpft.
Auf diese Weise entstehen aus den beiden DNA-Einzelsträngen der ursprünglichen Doppelhelix zwei neue und identische DNA-Doppelhelices.
Zu guter Letzt wollen wir uns noch ein schönes Bild aus der Wikipedia anschauen, das alles bisher Gesagte im Zusammenhang darstellt.
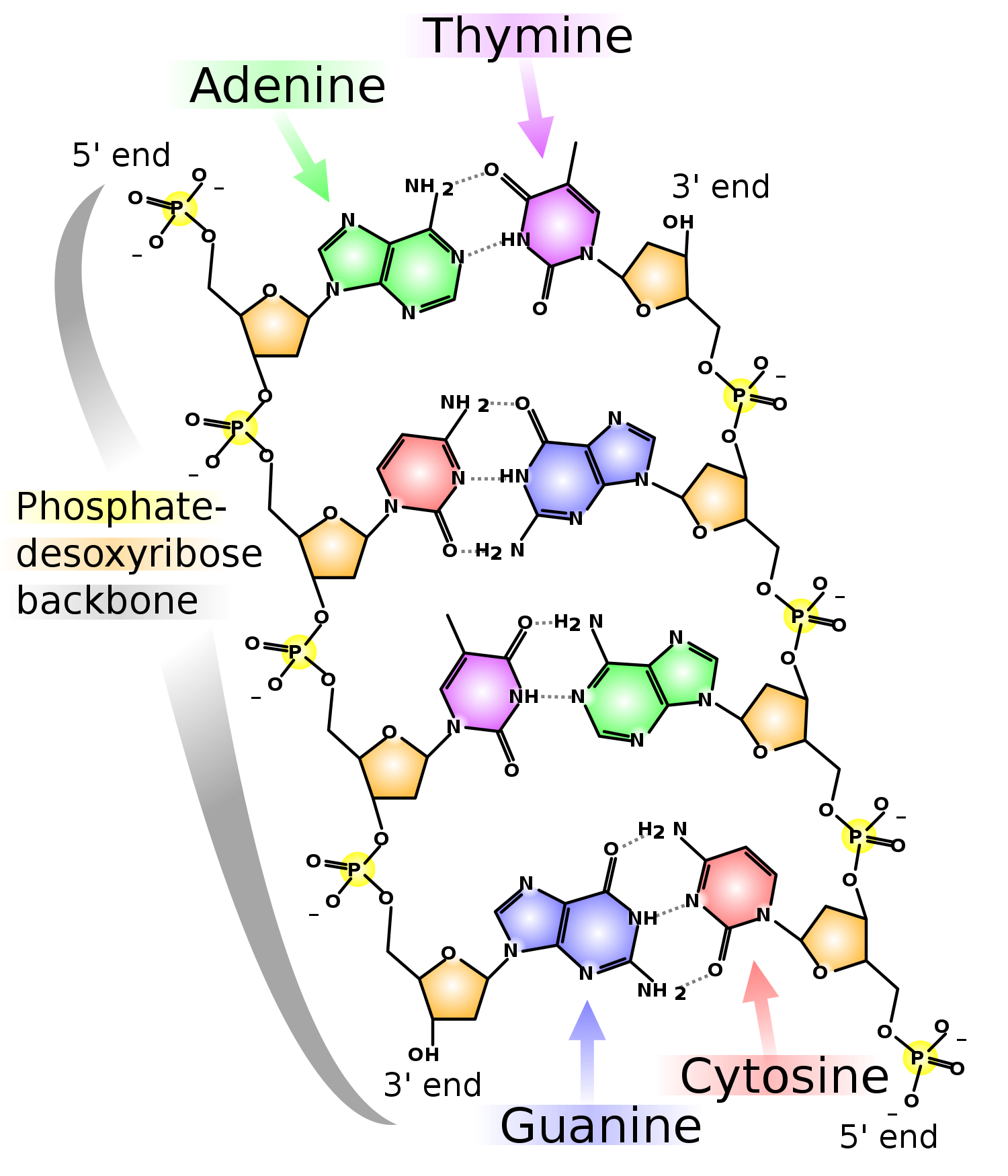
Ein winziger DNA-Doppelstrang aus vier Basenpaaren A-T, C-G, T-A und G-C.
Quelle: Wikipedia, Artikel "Basenpaar", Autor: Madeleine Price Ball (Madprime), Lizenz: Creative Commons Attribution-Share Alike 2.5 Generic, 2.0 Generic and 1.0 Generic license.
Auf diesem Bild sieht man eine Mini-DNA-Doppelhelix mit vier Basenpaaren. Achten Sie darauf, dass die beiden DNA-Stränge gegenläufig sind. Bei dem links dargestellten Strang ist das 3'-Ende unten und das 5'-Ende oben, bei dem rechten DNA-Strang ist es genau umgekehrt.
Würden die beiden DNA-Stränge in der gleichen Richtung verlaufen, was ja prinzipiell möglich wäre, dann hätte die DNA-Doppelhelix eine völlig andere, "verdrehte" oder "gekrümmte" Struktur. Andererseits würde die Replikation der DNA dann vielleicht wesentlich einfacher verlaufen, aber dazu kommen wir noch später.
Quellen:
- Oberstufen-Lehrbücher der Biologie
- Jochen Graw: Genetik, 7. Auflage, Springer Spektrum, Berlin 2021.
- Wikipedia, Artikel "Basenpaar".