Ein DNA-Stammbaum entsteht, wenn man die Basensequenzen bestimmter Gene (oft auch mitochondrialer Gene) verschiedener Arten von Lebewesen miteinander vergleicht und auswertet.
Die Grundidee bei einem DNA-Stammbaum
Im Laufe der allopatrischen oder sympatrischen Artbildung trennen sich Populationen von Lebewesen in zwei Teilpopulationen, die sich fortan unabhängig voneinander weiterentwickeln. In beiden Teilpopulationen treten immer wieder zufallsbedingte und nicht zielgerichtete Mutationen der DNA auf. Da der Genfluss zwischen den Teilpopulationen unterbrochen ist, führt das dazu, dass die DNA-Sequenzen der betrachteten Gene im Laufe der Zeit immer stärker voneinander abweichen. Man hat sogar festgestellt, dass es eine Art "molekularer Uhr" gibt: Die Zahl der Mutationen in der DNA ist abhängig von der vergangenen Zeit.
Vergleicht man also die Basensequenz eines bestimmten Gens in zwei verschiedenen Arten, dann kann man Unterschiede in der Sequenz feststellen. Je größer die Zahl der Unterschiede, desto länger existieren die beiden Arten unabhängig voneinander. Auf diese Weise kann man den ungefähren Zeitpunkt bestimmen, an dem sich die beiden Arten getrennt haben, als also noch der gemeinsame Vorfahr lebte.
Verfahren zur Erstellung eines DNA-Stammbaums
Das methodische Vorgehen, um einen DNA-Stammbaum zu erstellen, ist recht aufwendig und wird in dem Artikel "Sequenzstammbaum" im Spektrum-Lexikon der Biologie einigermaßen gut in vier Schritten erklärt [1]:
- Zunächst muss man nach homologen Genen suchen. Man kann nicht bei der Art A das Gen für Cytochrom C analysieren, und bei der Art B das Gen für die Alkoholdehydrogenase.
- Dann müssen die Basensequenzen der homologen Gene korrekt ausgerichtet werden.
- Der dritte Schritt ist die computergenerierte Erzeugung des Stammbaums selbst. Dieser Schritt ist recht komplex und Gegenstand der Bioinformatik.
- Im letzten Schritt muss der so erhaltene DNA-Stammbaum überprüft werden, dazu wird er meistens mit Stammbäumen verglichen, die mit Hilfe konventioneller Methoden (Fossilien, Ähnlichkeiten lebender Arten) erstellt worden sind.
Meistens stimmen die DNA-Stammbäume mit den morphologischen Stammbäumen überein, oft gibt es aber auch interessante Abweichungen oder gar Überraschungen.
Beispiele für DNA-Stammbäume
aus dem NRW-Abitur Biologie
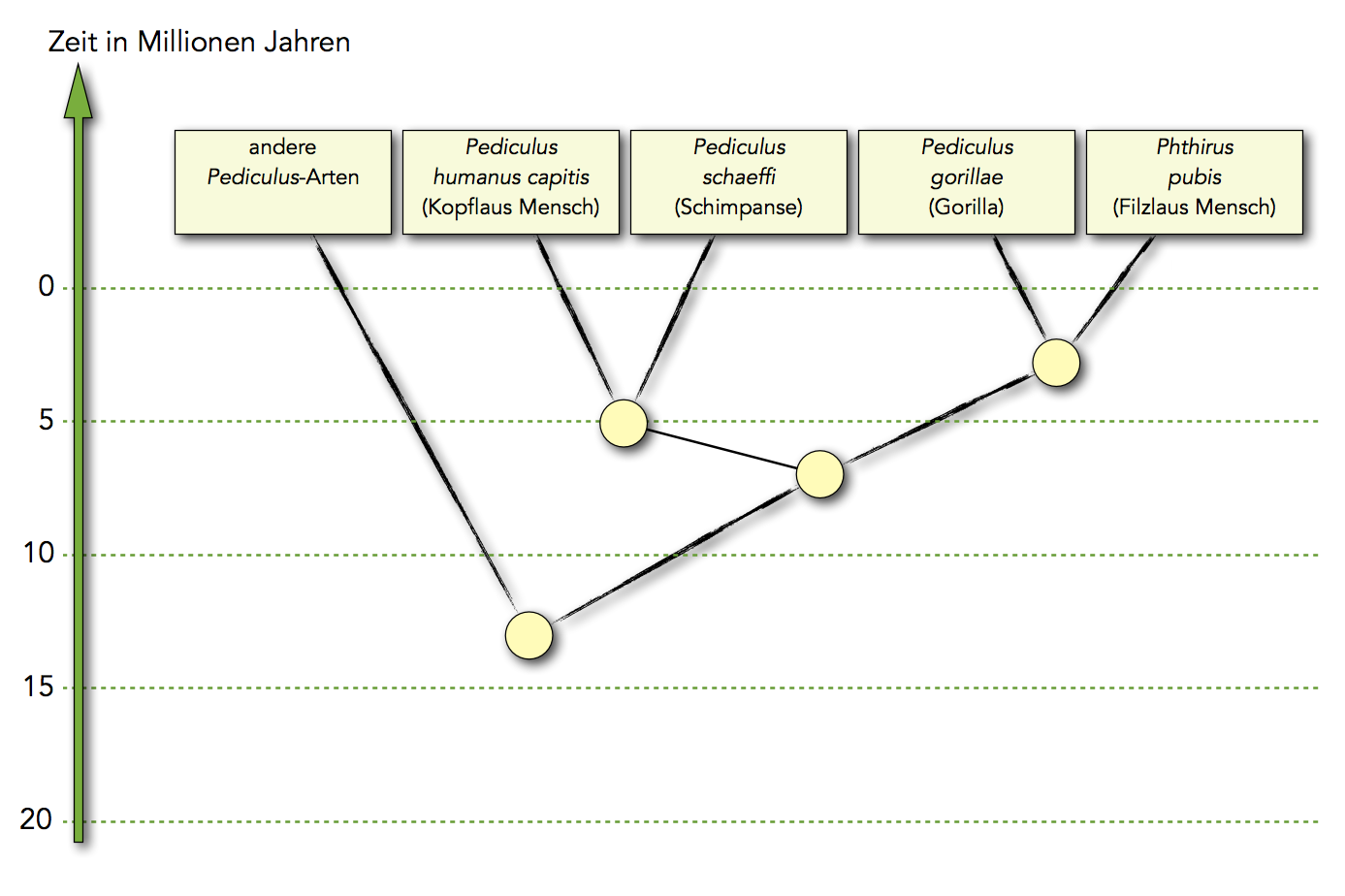
DNA-Stammbaum einiger parasitischer Läuse
Autor: Ulrich Helmich, Lizenz: Public domain
Das obige Bild zeigt den DNA-Stammbaum von Läusen, die als Parasiten auf Menschen und anderen Primaten vorkommen: Kopf- und Filzlaus des Menschen sowie eine Lausart, die im Fell von Schimpansen lebt und eine andere Lausart, die im Fell von Gorillas lebt. Die Daten zu diesem Stammbaum stammen übrigens aus der NRW-Abituraufgabe "Parasiten als Indikatoren der Primaten-Evolution" aus dem Jahre 2009. Zur Erstellung dieses Stammbaums wurden die Gene Cox1 und EF-1α analysiert [2]. In der LK-Abituraufgabe von 2013 "Stammesgeschichte des Chamäleons" ist ein DNA-Stammbaum verschiedener Chamäleon-Arten enthalten, und in der Abituraufgabe von 2015 finden wir einen DNA-Stammbaum von Großkatzen [2]:
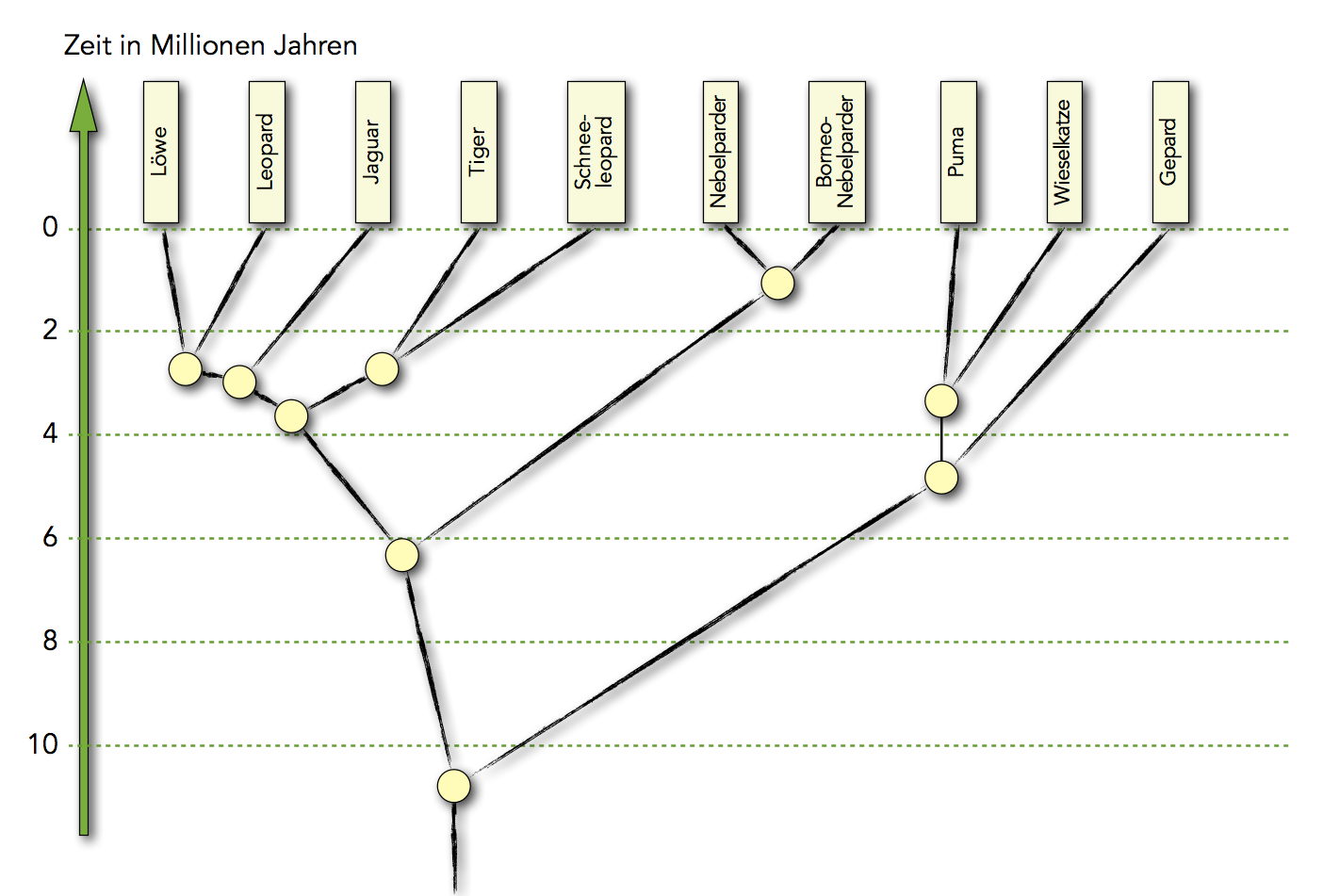
DNA-Stammbaum einiger Großkatzen
Autor: Ulrich Helmich, Lizenz: Public domain
Das Interessante an diesem Stammbaum: Man sollte doch eigentlich denken, dass der Löwe am nächsten mit dem Tiger verwandt ist, zumal diese beiden Arten sogar Hybriden bilden können, die sogenannten Liger. Die DNA-Analysen zeigen jedoch, dass der nächste Verwandte des Löwen der Leopard ist, die beiden Arten haben sich erst vor ca. 2,5 Millionen Jahren voneinander getrennt.
DNA-Stammbäume im NRW-Abitur
Das Thema "DNA-Stammbaum" bzw. "phylogentischer Stammbaum" ist für die Abiturienten unter Ihnen hochinteressant und wichtig. Auch in der NRW-Abituraufgabe "Die Evolution der Höhlenzikaden auf Hawaii" von 2016 [2] finden wir einen DNA-Stammbaum. Dieses mal wurde die DNA der Mitochondrien verschiedener Zikadenarten untersucht. In der Aufgabe "Die Evolution der giftigen Pitohuis auf Neuguinea", NRW 2017 [2], wurden die Verwandtschaftsverhältnisse der giftigen Pitohuis-Vögel mit Hilfe eines DNA-Stammbaums untersucht, auch hier wurde die mtDNA analysiert. Die Abituraufgabe "Wie die Fliege zum Kaktus kam – die Evolution einer Nahrungsbeziehung" enthält einen DNA-Stammbaum von vier Drosophila-Arten (Fruchtfliege) [2], und die Abituraufgabe "Molekulare Angepasstheit von Säugetieren an ein Leben im Wasser" von 2019 [2] enthält einen sehr umfangreichen DNA-Stammbaum ausgewählter Säugetierarten mit interessanten Erkenntnissen:
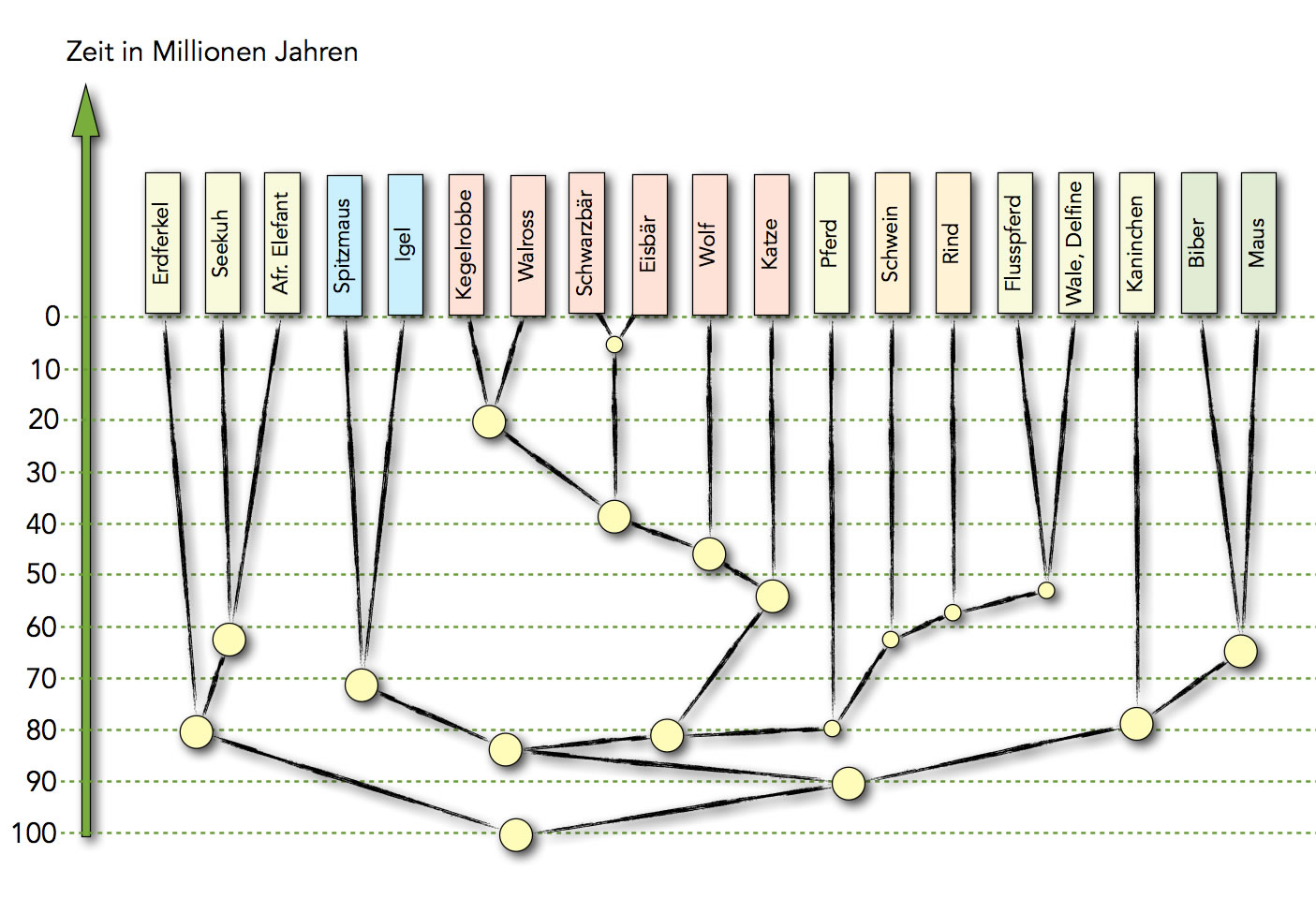
DNA-Stammbaum einiger Säugetiere
Autor: Ulrich Helmich, Lizenz: Public domain
Die Sehkuh ist zum Beispiel eng verwandt mit dem afrikanischen Elefanten, während sie nur entfernt verwandt mit dem Walross ist. Die Ähnlichkeit zwischen Seekuh und Walross ist auf konvergente Entwicklung wegen ähnlicher Umweltbedingungen zurückzuführen. Hasen und Kaninchen wurden früher zu den Nagetieren gerechnet, heute weiß man - auch aufgrund genetischer Untersuchungen - dass die eine eigene Ordnung innerhalb der Klasse der Säugetiere darstellen. Dass Spitzmaus und Maus nicht enger verwandt ist, wusste man schon früher, aufgrund morphologischer Merkmale. Der DNA-Stammbaum bestätigt diese Annahme in eindrucksvoller Weise.
Quellen:
- Spektrum-Lexikon der Biologie, Artikel "Sequenzstammbaum"
- Verschiedene Abituraufgaben, NRW Biologie