Eine Enzymreaktion wird untersucht
In einem Gedanken-Versuch werden acht Reagenzgläser mit Glucose-Lösungen unterschiedlicher Konzentration befüllt. Dann geben wir in jedes Reagenzglas je 10 Tropfen einer Lösung, die ein Enzym enthält, das Kohlendioxid aus Glucose freisetzt (Einzelheiten sparen wir uns hier, weil ich mir das Beispiel nur ausgedacht habe). Die folgende Tabelle zeigt nun die (hypothetischen) Versuchsergebnisse:
| c(Glucose) in mol/l | V(CO2) in μl / s |
| 0,01 | 1 |
| 0,02 | 4 |
| 0,05 | 12 |
| 0,1 | 21 |
| 0,2 | 34 |
| 0,5 | 59 |
| 1,0 | 75 |
| 2,0 | 78 |
Hier die graphische Darstellung der Werte (ein μl ist übrigens 1/1000 ml):
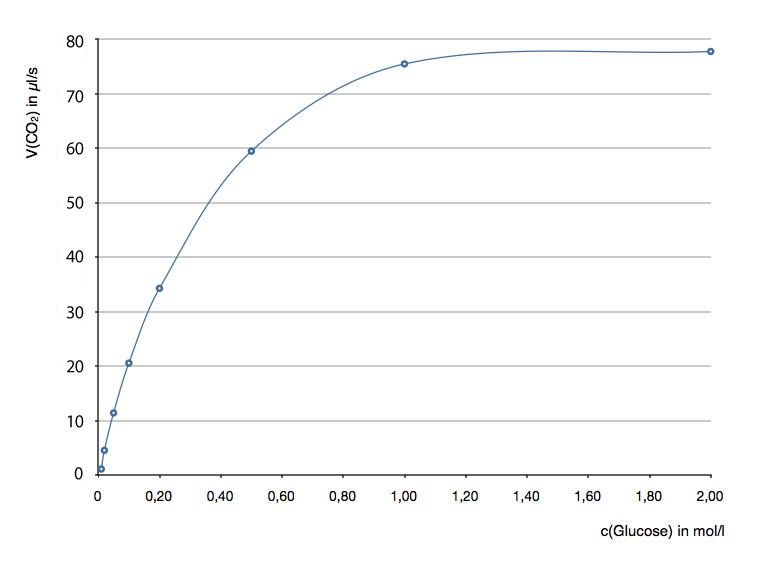
Graphische Darstellung der Versuchsergebnisse
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Diese Kurve sieht ganz nett aus, entspricht aber eigentlich gar nicht den Erwartungen, die man hat.
Für chemisch Vorgebildete:
Aus dem Chemieunterricht der Oberstufe wissen Sie vielleicht, das die Geschwindigkeit vR einer chemischen Reaktion von der Konzentration c(A) der Ausgangsstoffe abhängt.
Für monomolekulare Reaktionen, bei denen ein Ausgangsstoff A zu einem Produkt P reagiert, gilt die Gleichung
vR = k * c(A)
wobei k die Geschwindigkeitskonstante und c(A) die Konzentration des Ausgangsstoffs ist.
Bei bimolekularen Reaktionen, bei denen A-Teilchen und B-Teilchen zusammenstoßen müssen, damit P-Teilchen entstehen, gilt die Gleichung
vR = k * c(A) * c(B)
Auch bei der Zersetzung der Glucose sollte die Reaktionsgeschwindigkeit linear von der Glucosekonzentration abhängen:
vR = k * c(Glucose)
Die graphische Darstellung, in der die Abhängigkeit der Reaktionsgeschwindigkeit von c(Glucose) gezeigt wird, sollte also die Form einer Geraden haben, die durch den Nullpunkt des Koordinatensystems geht. Statt dessen sehen wir eine typische Sättigungskurve. Das kann man mit der "einfachen" Reaktionskinetik mono- oder bimolekularer Reaktionen (siehe Kasten für Experten) nicht erklären.
Aber natürlich kann man das Phänomen erklären, wenn man sich die Arbeitsweise eines Enzyms näher anschaut.
Erklärung
Ein Enzymmolekül kann pro Sekunde nur eine bestimmte maximale Zahl von Substratmolekülen umsetzen: die Enzymkapazität ist begrenzt. Stellen wir uns einmal vor, die Kapazität unseres Enzyms läge bei einer Wechselzahl von 9.000 Glucose-Molekülen pro Sekunde.
Unter der Wechselzahl versteht man die Zahl der Substrat-Moleküle, die ein Enzym-Molekül in einer bestimmten Zeit höchstens verarbeiten kann.
Angenommen, im ersten Reagenzglas (0,01 mol/l) trifft jedes Enzym-Molekül pro Sekunde im Schnitt auf 100 Glucose-Moleküle. Dies ist weit unter der Kapazität des Enzyms, es werden also alle 100 Substrat-Moleküle problemlos weiterverarbeitet.
Im zweiten Reagenzglas (0,02 mol/l) trifft jedes Enzym-Molekül auf ca. 200 Glucose-Moleküle. Auch dies ist weit unter der Enzymkapazität, daher können auch hier alle Glucose-Moleküle gespalten werden.
Auch bei den nächsten Reagenzgläsern ist dies der Fall: Die Enzyme treffen auf weniger Glucose-Moleküle pro Sekunde, als sie theoretisch verarbeiten könnten. Die Kapazität des Enzyms ist hier noch nicht ausgeschöpft.
Mit steigender Glucosekonzentration wird es aber langsam kritisch. In den letzten Reagenzgläsern ist die Glucosekonzentration schon so hoch, dass ein Enzym-Molekül nicht mehr jedes Glucose-Molekül verarbeiten kann, auf das es trifft. Irgendwann ist die Glucosekonzentration so hoch, dass alle Enzym-Moleküle unter Volllast arbeiten. Die Kapazitätsgrenze ist erreicht.
Wenn wir die Glucosekonzentration jetzt noch mehr erhöhen, verändert sich gar nichts mehr. Mehr als mit maximaler Kapazität können die Enzyme nicht arbeiten: 9.000 Glucosemoleküle pro Enzym und pro Sekunde. Und dann ist Schluss!
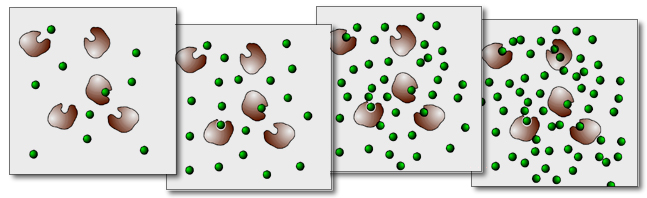
Veranschaulichung der Enzym-Kinetik
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
In dieser Abbildung ist das Ganze noch einmal anschaulich dargestellt. Im linken Kasten (niedrige Substratkonzentration) sind die Enzyme nicht ausgelastet. Manche Enzym-Moleküle sind sogar noch nicht von Substrat-Molekülen besetzt, sie "haben nichts zu tun". Im zweiten Kasten ist die Substrat-Konzentration zwar höher, aber die Enzyme arbeiten immer noch nicht mit Volllast. Selbst im dritten Kasten ist - zur Zeit der "Aufnahme" - noch ein Enzym-Molekül unbesetzt. Im vierten Kasten schließlich ist die Substratkonzentration so hoch, dass alle Enzyme voll ausgelastet sind. Eine weitere Erhöhung der Substratkonzentration würde jetzt keine Steigerung der Umsetzungsrate mehr nach sich ziehen.
Enzymaktivität
Die Enzymaktivität hängt von der Konzentration c(S) der Substrate ab: Je höher c(S), desto größer die Enzymaktivität, also die Geschwindigkeit, mit der das Enzym die Substrat-Moleküle umsetzt.
Allerdings ist diese Abhängigkeit nicht linear, sondern liegt in Form einer Sättigungskurve vor.
Ursache hierfür ist die begrenzte Kapazität eines Enzyms. Ein Enzym-Molekül kann pro Zeiteinheit nur eine bestimmte Anzahl von Substrat-Molekülen verarbeiten (Wechselzahl). Ist die Substratkonzentration zu hoch, sind die Enzyme voll ausgelastet. Eine weitere Steigerung der Substratkonzentration hat keine Steigerung der Enzymaktivität zur Folge; der Sättigungswert ist erreicht und kann nicht überschritten werden.
Michaelis-Menten-Kinetik
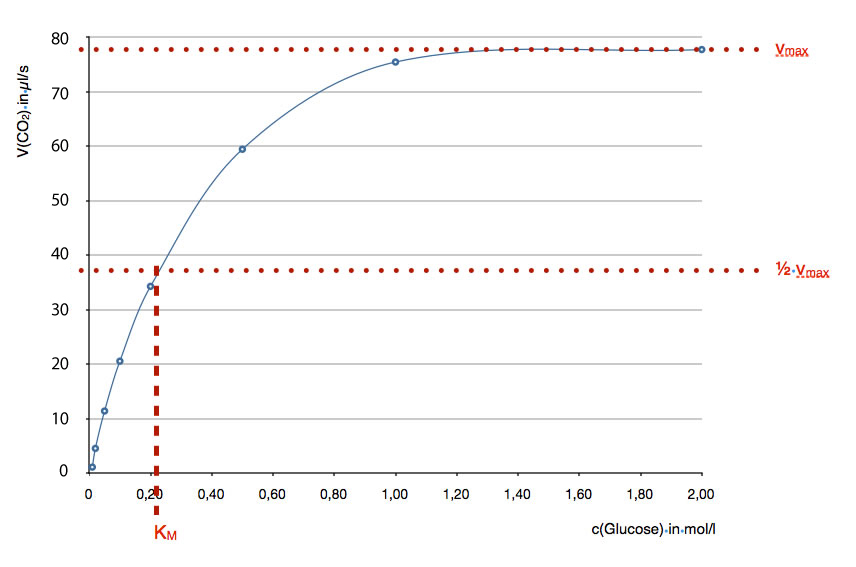
Eine typische Enzym-Kinetik
Autor: Ulrich Helmich 2016, Lizenz: siehe Seitenende.
Hier sehen wir noch einmal die gleiche Enzym-Kinetik wie in der Abbildung 1. Diese Abbildung soll zeigen, wie man die sogenannte Michaelis-Konstante KM graphisch ermittelt. KM ist ein quantitatives Maß für die Enzymaktivität.
Das Problem bei einer Sättigungskurve ist Folgendes: Man kann nicht ganz exakt sehen, bei welcher Glucose-Konzentration c(Glucose) der maximale Geschwindigkeit der Umsetzung erreicht ist. Ist die Geschwindigkeit von 78 µl CO2/s (oberer punktierte Linie) schon bei c(Glucose) = 1,40 mol/l erreicht oder erst bei 1,60 mol/l ?
Mit einem Geodreieck ist es aber kein Problem, zu einer genaueren Aussage zu kommen. Man kennt die maximale Geschwindigkeit vmax, in unserem Beispiel also 78 µl CO2/s. Davon nimmt man nun die Hälfte von vmax = 1/2 vmax, also 39 µl CO2/s und zeichnet in dieser Höhe eine weitere Linie.
An dem Punkt, wo die 1/2 vmax- Linie die Kurve schneidet, zeichnet man dann eine senkrechte Linie in das Diagramm ein. Dort, wo diese senkrechte Linie die waagerechte Achse des Diagramms schneidet, findet man den sogeannten KM-Wert. Und dann haben wir schon die Michaelis-Konstante KM mit 0,21 mol Glucose/l ermittelt.
Michaelis-Konstante KM = die Substratkonzentration, bei der das Enzym mit genau der halben maximalen Geschwindigkeit arbeitet.
Verschiedene Enzyme haben unterschiedliche KM-Werte
Evolution ist - knapp ausgedrückt - nichts anders als langsame schrittweise Anpassung an bestimmte Umweltbedingungen über viele, viele Generationen hinweg. Auch die Enzyme der Zellen haben sich im Laufe der Evolution an die üblichen Substratkonzentrationen angepasst, denen sie ausgesetzt sind. In einer "normalen" Umgebung wird ein Enzym nie mit seiner maximalen Kapazität arbeiten. Dann wäre ja gar keine "Luft mehr nach oben", falls sich die Substratkonzentration durch bestimmte Umstände einmal erhöhen sollte.
Die meisten KM-Werte passen zu der durchschnittlichen Substratkonzentration, der die Enzyme normalerweise ausgesetzt sind. Hier ein paar Beispiele:
Chymotrypsin hat einen KM-Wert von 5000 µmol, Carboanhydrase einen KM-Wert von 8000 µmol. Die Penicillinase hat einen geringen KM-Wert von 50, und die Arginin-tRNA-Synthetase setzt das Substrat Arginin mit einem KM-Wert von 3 um [3].
Diese Anpassung des Enzyms an die "übliche" Substratkonzentration hat den Vorteil, dass das Enzym sein Substrat unter Normalbedingungen rasch umsetzen kann. Auch eine leichte Erhöhung der Substratkonzentration kann das Enzym "verkraften", weil ja noch Kapazitäten frei sind. Wenn die normale Substratkonzentration dem KM-Wert entspricht, ist ja nur die Hälfte aller Enzym-Moleküle mit Substraten besetzt, die andere Hälfte "hat nichts zu tun" und kann auf Erhöhungen der Substratkonzentration reagieren.
Für Profis:
Arginin-tRNA-Synthetase ist ein Enzym, das bei der Translation eine wichtige Rolle spielt. Es belädt unter Verbrauch von ATP die transfer-RNA für Arginin, also ArgtRNA, mit der Aminosäure Arginin. Das Enzym setzt also drei Substrate um: Arginin, ArgtRNA und ATP. Man sollte nun ja denken, dass die KM-Werte für diese drei Substrate gleich groß sind. Das ist aber nicht der Fall. Der KM-Wert für Arginin liegt bei 3, der für ArgtRNA bei 0,4 und der für ATP bei 400 µmol [3].
Dieses Beispiel zeigt eindrucksvoll, dass ein- und dasselbe Enzym unterschiedliche KM-Werte für verschiedene Substrate besitzen kann.
Aufgabe
Zwei Enzyme dienen in der Leber zum Abbau der Glucose, nämlich Hexokinase und Glucokinase. Nun hat die Glucokinase aber einen 50 mal höheren KM-Wert als die Hexokinase.
Begründen Sie, welches Enzym hauptsächlich die Glucose abbaut, wenn die Glucose-Konzentration in der Leber sehr niedrig / sehr hoch ist.
Lösungsvorschlag:
Der KM-Wert der Hexokinase wird bereits bei sehr geringen Glucose-Konzentrationen erreicht. Die Hexokinase arbeitet also schon mit "halber Kraft", während die Glucokinase noch lange nicht ausgelastet ist. Sie würde erst bei einer 50fach höheren Glucose-Konzentration mit "halber Kraft" arbeiten. Bei niedrigen Glucose-Konzentrationen wird die Glucose also hauptsächlich durch die Hexokinase abgebaut. Bei einer hohen Glucose-Konzentration ist die Hexokinase dagegen völlig ausgelastet, und nun springt die Glucokinase ein, die mit wesentlich höheren Glucose-Konzentrationen zurecht kommt.
Für Profis:
Bei ständig wechselnden Substratkonzentrationen werden auch sogenannte Isoenzyme eingesetzt. Das sind Enzyme, die zwar unterschiedlich gebaut sind, aber trotzdem das gleiche Substrat umsetzen und die gleiche Reaktion katalysieren. Die eine Variante hat dann einen niedrigen KM-Wert und eignet sich daher gut für geringe Substratkonzentrationen, während die Iso-Variante einen höheren KM-Wert hat und somit besser für hohe Konzentrationen des Substrats geeignet ist. Oft haben die Isoenzyme eine fast identische Struktur.
Genetisch kann man das leicht mit einer Gen-Duplikation erklären. Das Gen für das ursprüngliche Enzyme wurde durch eine Chromosomenmutation dupliziert, also verdoppelt. Das Duplikat hat sich dann im Laufe der Evolution leicht verändert, so dass das neue Enzym eine etwas andere Aminosäuresequenz hat, was eine etwas andere Struktur des aktiven Zentrums zur Folge hat.
Quellen:
- Kompaktlexikon der Biologie, Spektrum-Verlag
- Nelson, Cox: LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
- Berg, Tymoczko, Gatto jr., Stryer: Stryer Biochemie, 8. Auflage, Springer Berlin Heidelberg 2018.
Seitenanfang -
Weiter mit
der Gibbs-Helmholtz-Gleichung ...