Ein Beispiel
Schon auf der Seite über die Sekundärstrukturen hatten wir folgendes Bild aus der Wikipedia betrachtet:
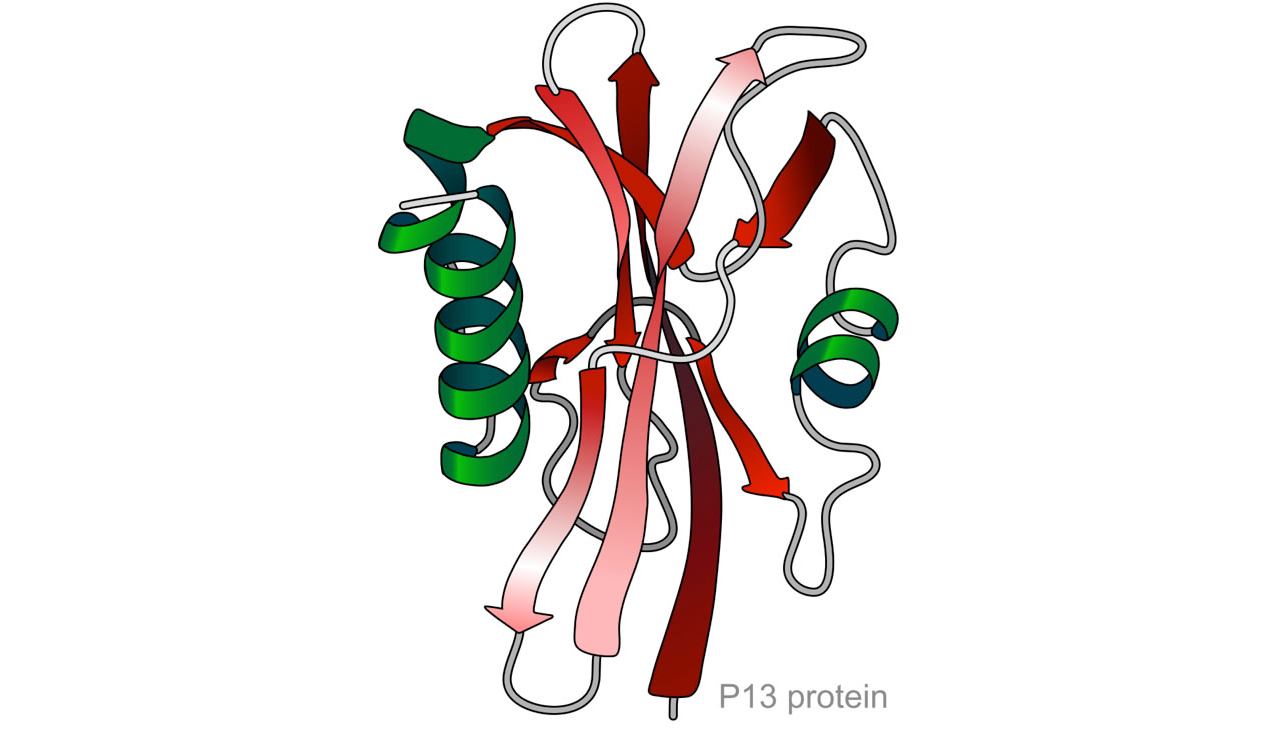
Die Tertiärstruktur eines kleinen Proteins
Quelle: Wikipedia, Artikel "Protein structure", abgerufen im Mai 2013, Autor: nicht mehr bekannt, Lizenz: public domain.
Dieses Bild zeigt die Tertiärstruktur eines kleinen Proteins. Die Tertiärstruktur ist die tatsächliche räumliche Anordnung der Aminosäuren, also die dreidimensionale Raumstruktur des Proteins. Im Prinzip wird die Tertiärstruktur mit ihren darin enthaltenen Sekundärstrukturen bereits von der Primärstruktur des Proteins vorbestimmt. Allerdings helfen auch bestimmte Enzyme, die sogenannten Chaperone dabei, die Tertiärstruktur herzustellen.
Was hält die Tertiärstruktur zusammen?
Die Tertiärstruktur wird durch vier verschiedene Arten chemischer Bindungen zusammengehalten. Nach abnehmender Stärke geordnet sind dies:
- Disulfidbrücken
- Elektrostatische Anziehungen
- Wasserstoffbrücken-Bindungen
- Hydrophobe Wechselwirkungen
Fangen wir mit den Disulfidbrücken an. Disulfidbrücken sind "richtige" starke kovalente Bindungen zwischen zwei nicht-benachbarten Aminosäuren.
1. Disulfidbrücken
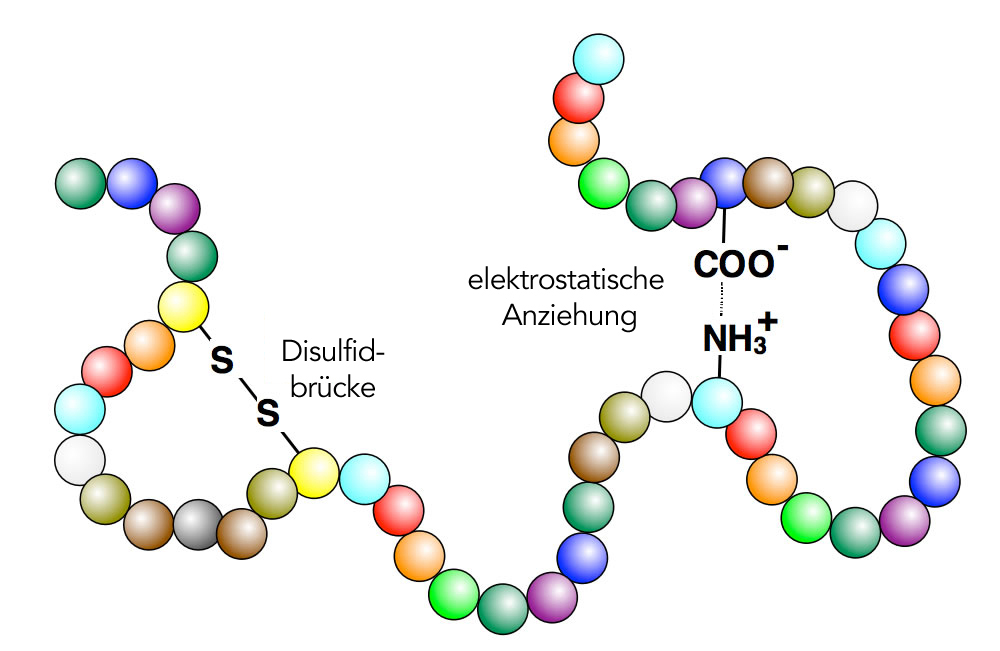
Disulfidbrücken und elektrostatische Anziehungen in einem Peptid
Autor: Ulrich Helmich, Lizenz: Public domain
Links im Bild sehen wir eine sogenannte Disulfidbrücke. Wie bereits auf der Seite über Aminosäuren ausgeführt, entstehen Disulfidbrücken zwischen zwei nicht-benachbarten Cystein-Resten in einem Protein:
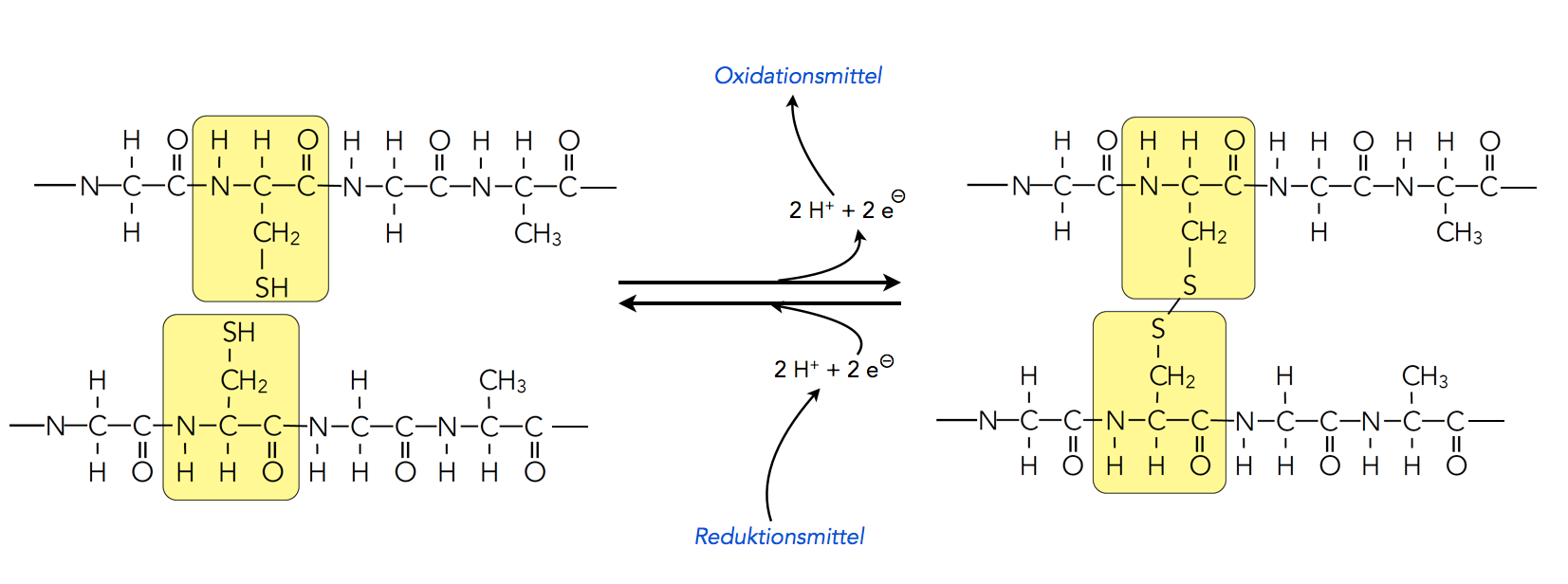
Bildung bzw. Spaltung einer Disulfidbrücke in einem Protein
Autor: Ulrich Helmich, Lizenz: Public domain
Damit eine Disulfidbrücke entsteht, muss ein Oxidationsmittel anwesend sein, das den freigesetzten Wasserstoff aufnimmt. Achtung: Bei der Bildung einer Disulfidbrücke werden nicht nur Protonen H+ abgegeben, sondern Protonen und Elektronen, was formal als 2H+ + 2 e- geschrieben wird. Es handelt sich also nicht um eine Säure-Base-Reaktion, sondern um eine Redoxreaktion.
Auch ist die Bildung von Disulfidbrücken durchaus reversibel. In Anwesenheit eines Reduktionsmittels, also eines Stoffes, der Wasserstoff (2H+ + 2 e-) freisetzen kann, kann eine Disulfidbrücke "gesprengt" werden, es entstehen dann wieder zwei Cystein-Reste.
Disulfid-Brücken und Dauerwellen
Disulfid-Brücken spielen beim Formen der Haare beim Friseur (oder wie sagt man heute - Hairstyler?) eine große Rolle. Die Haare werden zunächst in der gewünschten neuen Form fixiert (Lockenwickler), dann wird ein Reduktionsmittel wie beispielsweise β-Mercaptan oder Thioglycolsäureester dazugegeben [4], so dass sich ca. 25% der Disulfidbrücken in den Strukturproteinen der Haare lösen. Die feuchte Wärme, die während der Behandlung angewandt wird, hilft dabei, sie bricht Wasserstoffbrücken-Bindungen und elektrostatische Anziehungen innerhalb der Proteine auf.
Dann wird das Reduktionsmittel ausgewaschen und ein Oxidationsmittel wie Wasserstoffperoxid oder eine Bromat-Lösung dazugegeben [4]. Es bilden sich neue Disulfidbrücken. Durch diese neuen Disulfid-Brücken bleiben die Haare dann längere Zeit in der neuen Form (Dauerwelle).
2. Elektrostatische Anziehungen ↑
Wie Sie bereits wissen, gibt es saure und basische Aminosäuren. Die sauren Aminosäuren wie Asparaginsäure enthalten in der Seitenkette eine zweite COOH-Gruppe, die basischen Aminosäuren wie Lysin eine NH2-Gruppe. Carboxygruppen können ein Proton abgeben und sind dann negativ geladen, während Aminogruppen ein Proton aufnehmen können und dann positiv geladen sind. Positiv und negativ geladene Seitengruppen ziehen sich aber elektrostatisch an, so wie auf der Abbildung 2 auch dargestellt.
In vielen Schulbüchern werden solche chemischen Bindungen als Ionenbindungen bezeichnet. Das ist im Prinzip korrekt, aber sachlich vielleicht doch nicht ganz richtig. Wenn man den Begriff "Ionenbindung" hört, denkt man sofort an Salzkristalle wie Kochsalz NaCl. Zwischen den Natrium-Ionen und den Chlorid-Ionen bestehen in einem solchen Kristall sehr starke Anziehungskräfte, daher haben die meisten Salze auch sehr hohe Schmelzpunkte.
Die Anziehungskräfte zwischen einer positiv und einer negativ geladenen Seitenkette innerhalb eines Proteins sind keine Ionenbindungen in diesem Sinne. Diese Bindungen sind viel schwächer als eine Ionenbindung in einem Kochsalz-Kristall. Woran liegt das?
Ganz einfach: Das Protein befindet sich in einem wässrigen Medium. Jede Menge Wasser-Moleküle umgeben die geladenen Seitenketten der Aminosäuren und schirmen die positiven und negativen Ladungen ab. Die Anziehungskräfte zwischen den beiden Seitenketten sind also noch deutlich vorhanden, aber sehr viel schwächer als die zwischen den Ionen eines NaCl-Kristalls, wo keine Wasser-Moleküle sind. Daher sollte man lieber von elektrostatischen Anziehungen sprechen und nicht von Ionenbindungen. Im Lehninger werden diese Bindungen übrigens als "salt bridges" bezeichnet, also als "Salzbrücken" [6]. Dieser Begriff ist aber auch etwas irreführend, weil man dann gleich wieder an einen Kochsalzkristall denkt.
Elektrostatische Anziehungen und der pH-Wert
Elektrostatische Anziehungen sind empfindlich gegenüber Änderungen des pH-Wertes im Umgebungsmedium. Angenommen, der pH-Wert sinkt, weil die Protonenkonzentration steigt. Die deprotonierten negativ geladenen COO--Gruppen nehmen dann ein Proton auf, und die negative Ladung verschwindet. Die gegenüberliegende basische Aminosäuren mit der NH3+-Gruppe in der Seitenkette verliert dann ihren negativen Bindungspartner, die elektrostatische Anziehung bricht zusammen.
Wenn der pH-Wert umgekehrt steigt, weil die Protonenkonzentration ab- und die Hydroxidionenkonzentration zunimmt, geben die positiv geladenen NH3+-Gruppen ihr Proton ab und werden neutral. Die negativ geladenen COO--Gruppen haben dann keinen Bindungspartner mehr.
Diese pH-Empfindlichkeit ist auch der Grund dafür, dass die meisten Enzyme ein pH-Optimum haben, bei dem sie mit der größten Effizienz arbeiten. Ein Verdauungsenzym, das beispielsweise im Magen des Menschen bei sehr niedrigen pH-Werten optimal arbeitet, verliert im Dünndarm seine Aktivität. Im Dünndarm herrscht ein leicht alkalisches Milieu, die positiv geladenen NH3+-Gruppen geben ihr Proton ab, werden neutral, und die elektrostatischen Kräfte, die vielleicht auch das aktive Zentrum des Enzyms stabilisiert haben, verschwinden.
3. Wasserstoffbrücken-Bindungen ↑
Seitenketten mit Carboxyl-, Hydroxyl- oder Aminogruppen, wie sie in einigen Aminosäuren vorkommen, können untereinander Wasserstoffbrücken-Bindungen eingehen. Auch dadurch wird die Tertiärstruktur eines Protein stabilisiert. Diese H-Brücken sind allerdings recht schwach und anfällig gegen Schwankungen der Temperatur und des pH-Wertes.
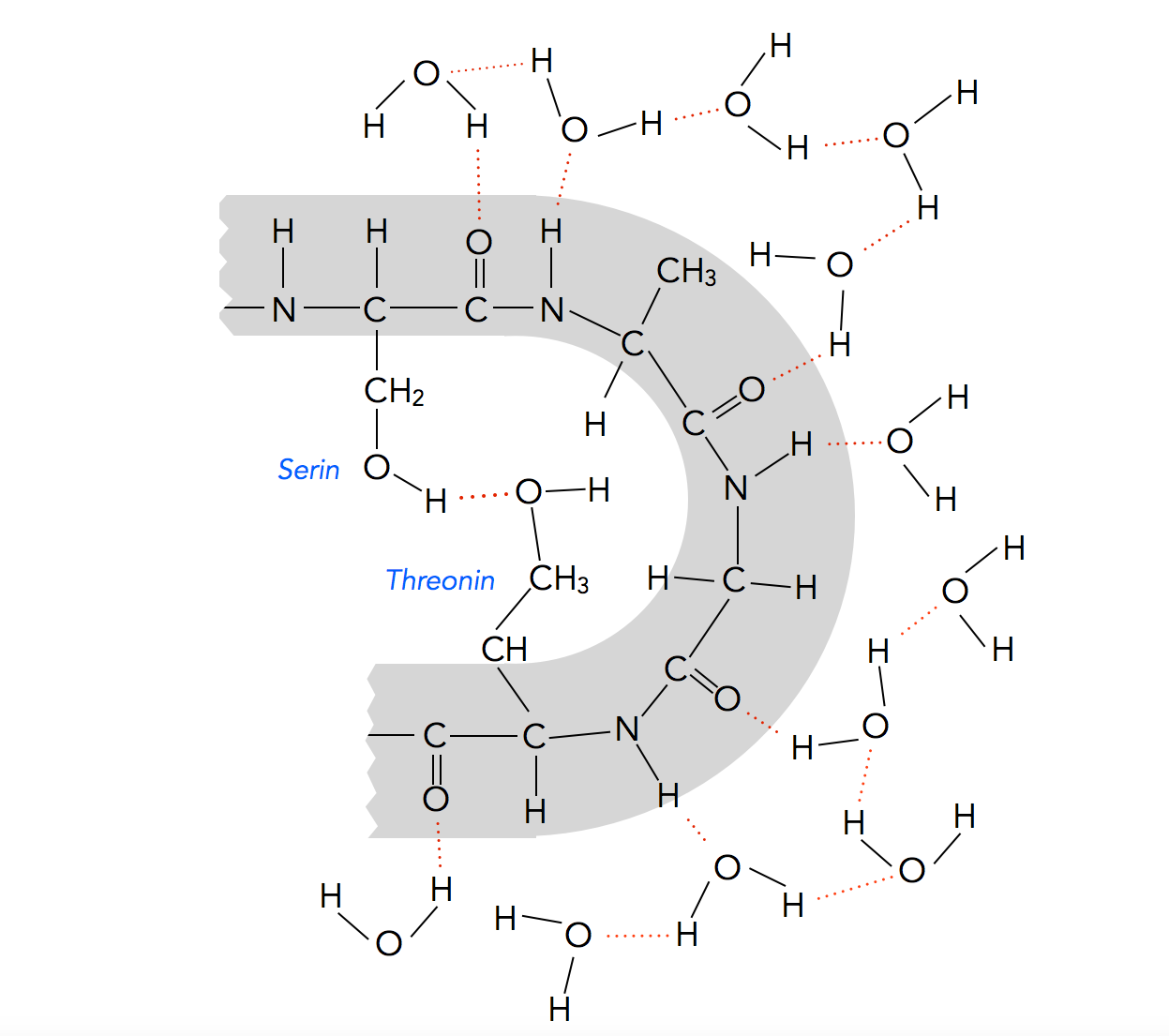
H-Brücken zwischen Seitenketten nicht-benachbarter Aminosäuren (Serin, Threonin)
Autor: Ulrich Helmich, Lizenz: Public domain
Eine H-Brücke allein beeinflusst die Tertiärstruktur eines Peptids noch nicht spürbar, aber wenn viele H-Brücken gebildet werden oder wenn sie mit anderen schwachen chemischen Bindungen kombiniert werden, hat das schon einen deutlichen Effekt auf die Tertiärstruktur eines Peptids oder Proteins. Immerhin sind H-Brücken die "stärksten der schwachen" chemischen Bindungen.
Auf dieser Seite des Chemie-Lexikons erfahren Sie mehr über H-Brücken.
4. Hydrophobe Wechselwirkungen ↑
Wenn man Öl in Wasser gießt und kräftig schüttelt, bildet sich zunächst eine Emulsion aus vielen kleinen Öltropfen, die im Wasser schwimmen. Nach kurzer Zeit aber vereinigen sich diese Öltropfen zu einer großen Ölschicht, die auf dem Wasser schwimmt. Ursache für diese Entmischung ist nicht etwas der starke Zusammenhalt der Öl-Moleküle, sondern der starke Zusammenhalt der Wasser-Moleküle. Die Wasser-Moleküle schließen sich zu einem Netzwerk zusammen, das von Wasserstoffbrücken zusammengehalten wird. Jedes H2O-Molekül bildet im Schnitt H-Brücken zu 3,4 anderen H2O-Molekülen aus, es bildet sich also in flüssigem Wasser ein dichtes und starkes Netzwerk.
Die Öl-Moleküle können keine H-Brücken zu Wasser-Molekülen ausbilden, können also an diesem Netzwerk nicht teilnehmen. Im Gegenteil, sie würden dieses Netzwerk sogar stören. Denn dort, wo sich Öl-Moleküle im Wasser befinden, können die Wasser-Moleküle keine H-Brücken mehr ausbilden. Das Wasser-Netzwerk würde dadurch instabiler werden.
Vereinfachend könnte man sagen, dass die Wasser-Moleküle mit den Öl-Molekülen "nichts zu tun haben wollen", und so bleibt den ausgeschlossenen Öl-Molekülen sozusagen nichts anderes übrig, als sich ihrerseits zusammen zu schließen. Van-der-Waals-Kräfte stabilisieren dann den Verband aus hydrophoben Molekülen bzw. Atomgruppen.
Auf dieser Seite des Chemie-Lexikons erfahren Sie mehr über den hydrophoben Effekt. Vor allem wird hier sehr genau erläutert, wie das H2O-Netzwerk auf das Vorhandensein von störenden lipophilen Molekülen reagiert.
Ähnlich ist es bei einem Peptid. Betrachten wir dazu die folgende Abbildung:
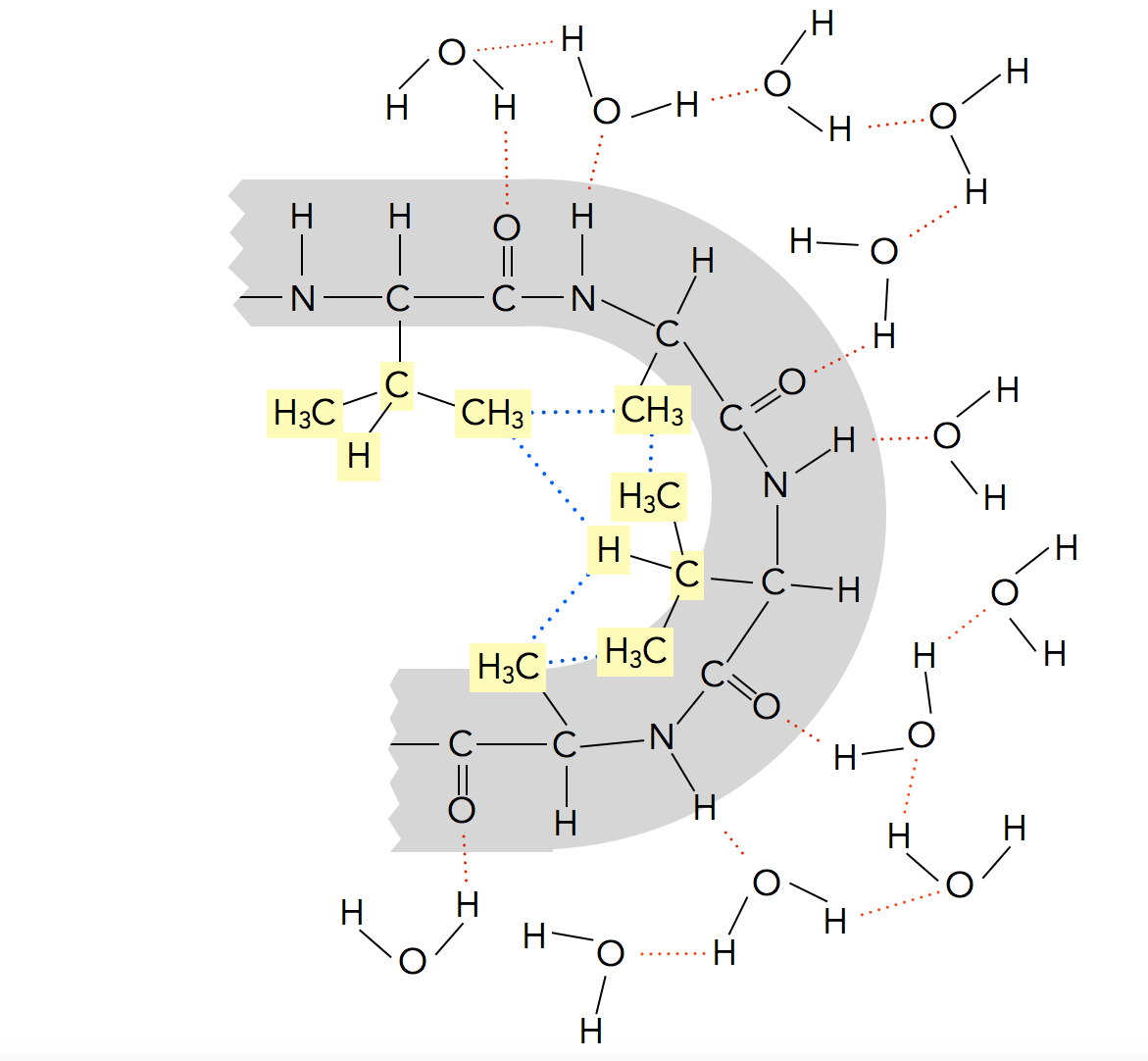
Auswirkung des hydrophoben Effekts auf die Proteinstruktur
Autor: Ulrich Helmich, nach einem Bild aus [2], Lizenz: Public domain
Dieser Ausschnitt aus einer Peptidkette besteht aus ungeladenen und unpolaren Aminosäuren wie Glycin, Alanin und Valin. Die Seitenketten dieser Aminosäuren sind hydrophob, "vermeiden" also den Kontakt mit Wasser-Molekülen. Die Folge einer solchen Anhäufung unpolarer Aminosäuren ist eine Krümmung der Peptidkette, so dass die unpolaren Seitenketten im Innern der Krümmung vor den Wasser-Molekülen weitgehend verborgen sind. Die H-Brücken zwischen den O- und N-Atomen der Peptidbindungen und den umgebenden Wasser-Molekülen sind rot eingezeichnet, die van der Waals-Kräfte zwischen den hydrophoben Seitenketten der Aminosäuren blau.

Das Tetrapeptid Ala-Ala-Val-Ala als Molekülmodell
Autor: Ulrich Helmich, Lizenz: Public domain
Dieses selbstgebaute Molekülmodell eines hydrophoben Tetrapeptids (Ala-Ala-Val-Ala) soll den hydrophoben Effekt noch einmal anschaulich verdeutlichen. Die hydrophoben Alkylreste der vier Aminosäuren sammeln sich in der "Mitte" des zusammengekrümmten Peptids.
Proteindomänen
Jane Richardson entdeckte 1981, dass es eine weitere Hierarchieebene zwischen Sekundärstrukturen und der Tertiärstruktur gibt. In vielen Proteinen tauchen immer wieder geordnete Bereiche auf, die sich aus mehreren alpha-Helices und/oder beta-Faltblättern zusammensetzen und die eine bestimmte Aufgabe innerhalb des Proteins übernehmen. Richardson nannte diese Bereiche "Domänen".
Wenn Sie mehr über die Proteindomänen lesen wollen, gehen Sie bitte auf diese Lexikon-Seite.
AlphaFold
Seit einigen Jahren gibt es eine komplexe Software, mit deren Hilfe man die Tertiärstruktur von Aminosäuresequenzen berechnen kann. "Die KI-Software AlphaFold2 bewältigt diese Aufgabe mit nie zuvor erreichter Genauigkeit, indem sie die Moleküle mit Hilfe maschinellen Lernens modelliert." [5]. Die Erfolgsquote von AlphaFold2 ist beeindruckend, obwohl durchaus manche Vorhersagen völlig daneben liegen, wenn man sie mit experimentellen Daten vergleicht. Inzwischen hat man der Software auch das "Halluzinieren" beigebracht, indem man eine gewünschte Tertiärstruktur vorgibt und die Software dann verschiedene Aminosäuresequenzen ausprobieren lässt.
Quellen:
- Römpp Chemie-Lexikon, 9. Auflage 1992
- Alberts, Bruce et al. Molekularbiologie der Zelle, 6. Auflage, Weinheim 2017.
- Stryer, Biochemie
- Chemgapedia: Die Chemie der Dauerwelle. Leider nicht mehr verfügbar.
- Callaway: "Revolution in der Proteinforschung". Spektrum der Wissenschaft 9/2022, S. 56ff.
- Nelson, Cox. LEHNINGER Principles of Biochemistry. Macmillan Learning, New York 2021.
Seitenanfang -
Weiter mit der Beeinflussung der Tertiärstruktur ...
Weiter mit Proteindomänen...